Adiciones sobre el micovirus Penicillimonavirus gammaplasmopara o Penicillimonavirus gammaplasmoparae (Autora: Ana B. Ruiz-García)
Si queréis tener las secuencias de los dos RNA del virus podéis bajárosla de GenBank con los números de Acceso OP042367 y OP042368.
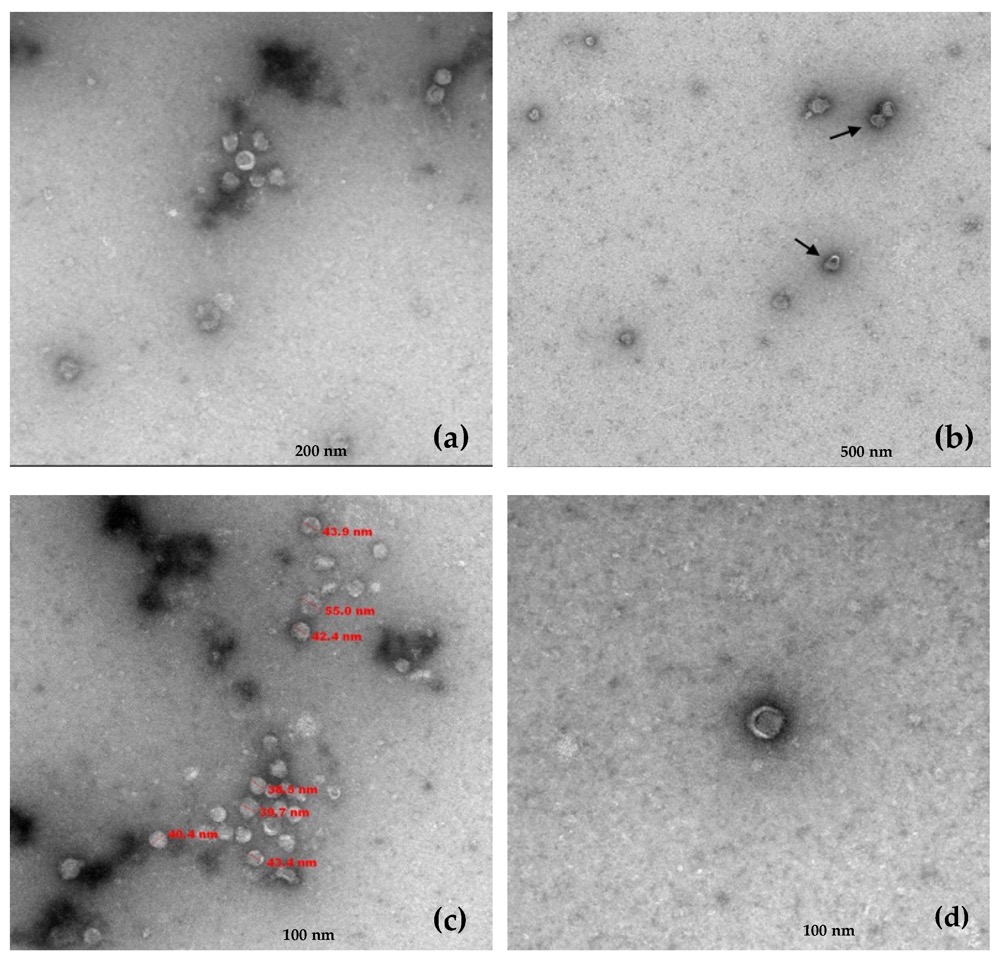
Para más información sobre el micovirus ahora podéis consultar nuestra publicación sobre los avances que hemos realizado en el artículo científico publicado en Plants. En el artículo podréis comprobar cómo es el virus cuando se observa por microscopio electrónica.
Desarrollamos y validamos un nuevo duplex para la identificación del viroide latente del melocotonero en colaboración con el EURL de Virus (Autora: A.B. Ruiz-García)
El control de este viroide sigue siendo problemático porque la mayoría de las variantes de PLMVd son asintomáticas y, aunque hay disponibles diversas técnicas de detección, la fiabilidad de los métodos basados en PCR se ve comprometida por la estructura secundaria compleja y ramificada de su ARN y por su diversidad genética.
En este estudio, se ha desarrollado y validado un método de RT-qPCR que potencialmente puede detectar todas las variantes conocidas de PLMVd. Además, para simplificar la preparación de la muestra, se ha puesto a punto un procedimiento rápido de extracción basados en el uso de extracto crudp o impresión de tejido. El rendimiento de este nuevo procedimiento se ha evaluado en un estudio interlaboratorio en el que han participado cinco laboratorios especializados.
Se ha demostrado que el nuevo método, en combinación con enfoques de preparación rápida de muestras, es factible y fiable, con la ventaja de detectar todos los diferentes aislados/variantes de PLMVd analizados en una sola reacción, lo que reduce los costos del diagnóstico de rutina.
Para detalles podeis consultar nuestro artículo publicado en la revista Plants “Development and Validation of a Duplex RT-qPCR for Detection of Peach Latent Mosaic Viroid and Comparison of Different Nucleic-Acid-Extraction Protocols”
Revisión del nuevo estándar de la EPPO para el diagnóstico de CTV (Autora: A. B. Ruiz-García)
Para más información podéis consultar la publicación PM7 31(2) Citrus tristeza virus
Desarrollamos el método más inclusivo para la detección del virus del enrrollado 2 (Autora: A.B. Ruiz-García)
Para más detalles os invitamos a leer el artículo publicado en la revista Plants con la descripción del trabajo
“A Novel and Highly Inclusive Quantitative Real-Time RT-PCR Method for the Broad and Efficient Detection of Grapevine Leafroll-Associated Virus 1”
Descubrimos el viroide del hoyuelo de la manzana (ADFVd) en granado (Autora: A.B. Ruiz-García)
Este estudio se inició cuando se recibieron en el laboratorio muestras con una sintomatología muy particular, tal como se muestra en la figura de abajo.
Figura 1. Granada con síntomas de anillos cloróticos. Muestra infectadas por el viroide del hoyuelo de la manzana - apple dimple fruit viroid (ADFVd).

Para la identificación del agente etiológico del posible agente causal de esta sintomatología se realizó secuenciación masiva a partir del ARN total de la muestra.
Los resultados mostraron la presencia del viroide del hoyuelo del manzano.
Al realizar prospecciones se observaron frutos con esta sintomatología como se muestra en la figura 2
Figura 2

La presencia del viroide se puede confirmar mediante RT-PCR con los iniciadores que diseñados en el estudio.
ADFVd F3: 5′-TTGACTAGATGCCCGCCTGAC-3′ y ADFVd R3 5′-CACGACCTAGAGCCGCCTCCA-3′ or ADFVd F4: 5′-AAGGACTTGACTAG-ATGCCCGCCTG-3 y ADFVd R4: 5′ AGTCATTAGCGCAGGGGGGTGC-3′.
Estudios de HTS revelan la diversidad del virus del mosaico del pepino (CMV) en Eslovaquia y nuevos huéspedes de CMV de la familia Papaveraceae (Autora: A.B. Ruiz-García)
Mediante secuenciación masiva, hemos caracterizado genomas completos de 14 variantes de CMV de diferentes plantas hospedadoras. De estas, tres variantes procedían de especies de Papaveraceae (amapola oleaginosa, amapola común y celidonia mayor), previamente poco descritas como huéspedes naturales del CMV. Los aislados de CMV estudiados y que eran de Eslovaquia se pueden dividir en dos grupos genéticamente diferentes, Ia y II, respectivamente.
La variante SL50V, caracterizada por una secuencia de ARN2 divergente, representa potencialmente una variante recombinante.
En cuatro muestras (T101, SL50V, CP2, MVU2-21), se identificó la presencia de ARN de CMV satélite junto con CMV. Aunque se transmitió mecánicamente a plantas de pepino experimentales, no se pudo establecer el papel del ARN satélite en la sintomatología observada debido a una infección compleja de los huéspedes originales con diferentes virus.
Para más detalles podéis consultar el artículo “High-Throughput Sequencing Discloses the Cucumber Mosaic Virus (CMV) Diversity in Slovakia and Reveals New Hosts of CMV from the Papaveraceae Family”

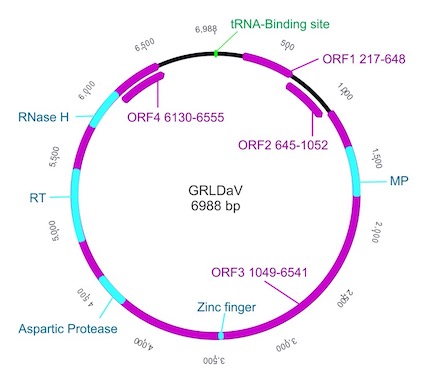
Caracterizamos por primera vez un micovirus asociado al oidio de la vid y averiguamos que consta de un genoma bipartito (Autores: A.B. Ruiz-García y A. Olmos)
Entre otras cosas hemos presentado por primera la secuencia completa del virus Plasmopara-viticola-lesion-associated mononegaambi virus 3, que representa la primera especie de un nuevo género para la que hemos determinado su estructura genómica que ha resultado ser bipartita.
De hecho en el laboratorio hemos identificamos el virus Plasmopara-viticola-lesion-associated mononegaambi virus 3, un mymonavirus asociado a hongos, en plantas de vid que muestran una sintomatología inusual de curvatura hacia arriba en las hojas y un deterioro prematuro.

La familia Mymonaviridae es una familia que comprende virus de ARN monocatenario de sentido negativo que infectan hongos filamentosos, que se han asociado a oomicetos, plantas e insectos.
Aunque la primera descripción del genoma de mymonavirus se realizó hace una década, la organización y estructura del genoma los géneros de la familia, no ha está clara hasta la fecha.
En el laboratorio hemos determinado el genoma completo del mymonavirus Plasmopara-viticola-lesion-associated mononegaambi virus 3 que representa la primera secuencia genómica completa de un posible nuevo género.
Además, proporcionamos pruebas sólidas de que el genoma de es bipartito y comprende dos moléculas de ARN de alrededor de 6150 y 4560 nt.
Nuestros resultados indican que el patógeno oídio, Erysiphe necator, también está presente en las plantas analizadas y sugieren que este virus puede estar infectando este hongo.
Sin embargo, aún está por determinar si el hongo y/o el micovirus están asociados con la sintomatología que inicialmente motivó estos estudios.
Estudios en el viroma del olivo en España, el olivavirus OLYaV (Autora: A.B. Ruiz-García)
En la figura 1 se muestra la sintomatología observada en campo en árboles de olivo.

Figura 1. Síntomas de defoliación, muerte y amarilleamiento de hojas causados por OLYaV.
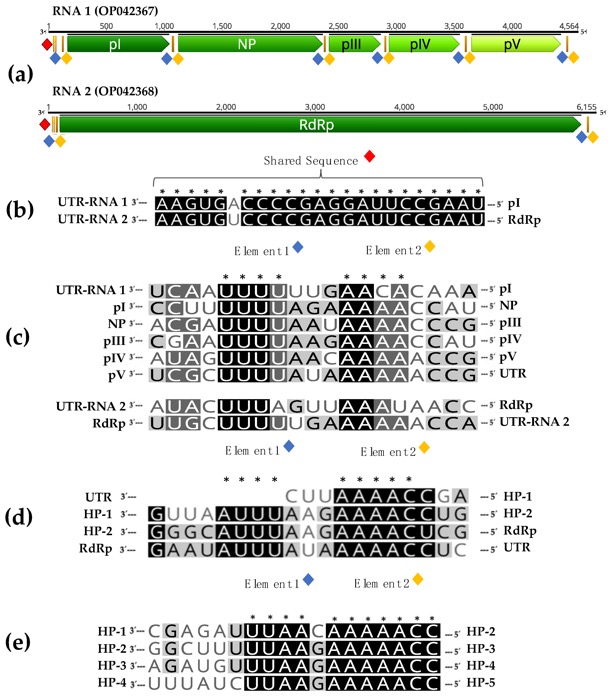
La comparación entre las secuencias nucleotídica/proteínica del nuevo aislado español de OLYaV y las secuencias nucleotídica/proteínica del aislado brasileño previamente secuenciado por nuestro equipo también, se muestra en la figura 2.
Como se puede observar las regiones más óptimas para la detección del virus deberán basarse en la región 5’UTR, p21 y/o 3’UTR y no la HSP70 que es la que actualmente se emplea y tiene un bajo porcentaje de homología (79.8%) lo que implica la presencia de falsos negativos porque la técnica puede no cubrir la variabilidad del virus.
Además se ha obtenido la segunda secuencia completa de OLYaV. Este virus también se ha detectado en un posible vector, el psílido Euphyllura olivina. Además, se ha confirmado en España también la presencia de Olea europaea geminivirus (OEGV), recientemente informado en Italia, y se ha obtenido la secuencia completa de dos aislados por secuenciación HTS y por secuenciación Sanger.
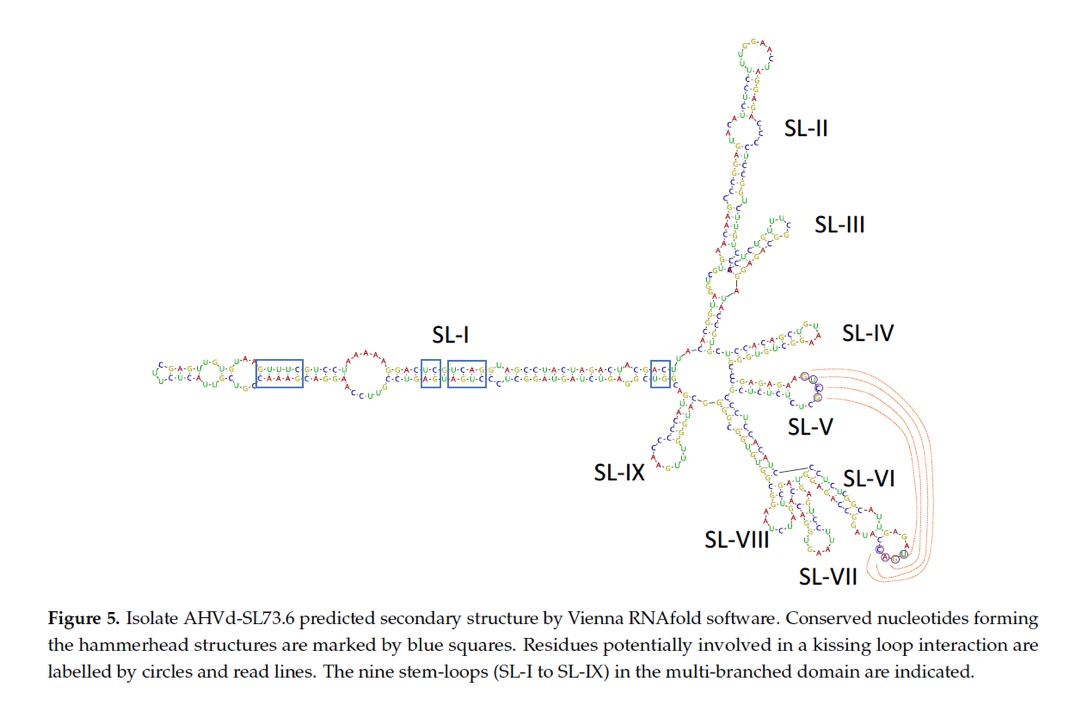
El níspero como nuevo huesped del viroide del manzano de cabeza de martillo (Autora: A.B. Ruiz-García)
En la siguiente figura se observa la estructura secundaria del viroide aislado de níspero.
Primeras detecciones de la falsa sharka en níspero en España (Autora: A.B. Ruiz-García)
La secuencia y caracterización genómica del primer aislado español de ACLSV infectando níspero se realizó de la siguiente forma.
El análisis por secuenciación masiva (HTS) realizado en el ARN total extraído de la muestra SL73.6 rindió 31.867.726 lecturas d con un tamaño promedio de 134,8 nt. Se realizó la sustracción del genoma del níspero, tras el control de calidad de las lecturas y eliminar los adaptadores, lo que resultó en 1.698.300 lecturas que se utilizaron para el ensamblado de novo que dio lugar a 13.797 contigs. Se anotaron los contigs relacionados con virus
y viroides mediante BLASTN/X. Este análisis mostró 11 contigs de tamaños entre
6511 y 281 nt relacionados con ACLSV, confirmando la presencia de ACLSV en la muestra. La extensión de los contigs realizando el mapeo de las lecturas contra los contigs permitió la recuperación de un genoma casi completo de 7533 nt, cubierto por 44 039 lecturas (cobertura promedio 857,2x), aislado llamado SL73.6 y depositado en GenBank, número de acceso OK272502. Este aislado mostró el mayor porcentaje de identidad a nivel nueclotídico (83,7%) con el aislado alemán 38/85-B (KX579123) de manzano. El mapa de la organización del genoma SL73.6, la cobertura promedio de HTS y la similitud de proteínas de sus ORF con el aislado de ACLSV más cercano (KX579123) se muestran en la Figura1
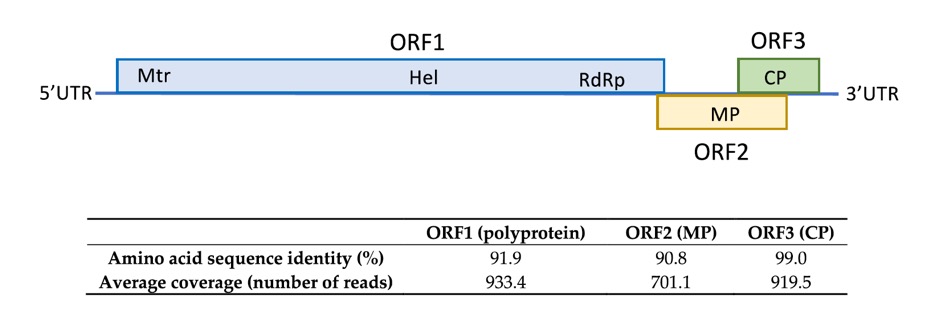
Figura 1. Similaridad aminoacídica del aislado SL73.6 con los ORFs del aislado de ACLSV más próximo (KX579123). Se muestra la cobertura por HTS.
En la siguiente figura se muestra la relación filogenética del aislado de ACLSV de níspero con otros aislados de ACLSV de otros orígenes y de otros hospedados.

Primeras detecciones de apple stem grooving virus en níspero en España (Autora: A.B. Ruiz-García)
La secuencia y caracterización genómica del primer aislado español de ASGV infectando níspero se realizó de la siguiente forma.
Se analizó por secuenciación masiva la muestra SL73.32 que dio como resultado 52.353.872 lecturas (con un promedio de tamaño de 134,34 nt). Se realizó el control de calidad con el recorte de adaptadores y de secuencias que no tenían la calidad suficiente. Las lecturas se mapearon contra el genoma de níspero para la sustracción del genoma del huésped y las 566.878 lecturas no relacionadas se sometieron a ensamblaje de novo, generando 519 contigs. Entre ellos, 7 contigs estaban relacionados, mediante análisis BLASTN/X, con el virus ASGV y tenían unos tamaños que oscilaron entre 1523 nt y 310 nt . El remapeo de las lecturas contra los contigs permitió la superposición entre algunos de los contigs y la recuperación de 3 secuencias parciales de 730, 1260 y 4291 nt que cubren el 96,7 % del genoma, faltando 98 nt en el extremo 5 prima; dos pequeñas regiones de codificación de 33 nt y 37 nt en el ORF1; y 48 nt en el extremo 3 prima, respecto a la secuencia de referencia (NC_001749). Para cubrir las dos brechas de ORF1, se realizaron RT-PCR y secuenciación de Sanger. La superposición entre las secuencias HTS parciales y las secuencias amplificadas por RT-PCR resultó en el ensamblaje de una secuencia de codificación ASGV de longitud casi completa de 6345 nt, SL73.32 (depositado en GenBank, número de acceso OK272504). SL73.32 mostró el mayor porcentaje de identidad de nucleótidos (95,1 %) con el aislado de cítricos FKSS2 (LC143387) de Japón y una similitud de nucleótidos del 83,42% con el aislado de níspero L3 de China (MK599422). Estos resultados confirman la aparición de ASGV en nísperos en España. El mapa de la organización genómica de SL73.32, cobertura promedio de HTS y similitud de proteínas de sus ORF con el aislado de ASGV más cercano (LC143387) se muestra en la Figura 1.

El estudio filogenético del aislado viral de níspero español con otros aislados en otros cultivos y con los aislados ASGV de cítricos (también denominados CTLV) se muestra en la Figura 2

Primera detección en España de PeSV en granado (Autora: A.B. Ruiz-García)
La extensión de los contig se realizó mediante el programa Geneious Prime y permitió recuperar una secuencia genómica de PeSV de 9926 nt casi completa (MZ361583, cobertura promedio 528x). Esta secuencia mostró una identidad de nucleótidos del 78,1 al 80,8 % en comparación con las secuencias genómicas de PeSV (MT680930-MT680935). No se detectó ningún otro virus en el análisis HTS.
La RT-PCR de la planta de granado original con los cebadores diseñados en este trabajo SPeSV-6F (5'-GGCTAGAAACGGTGGGATGA-3') y SPeSV-6R (5'-ACCACCTGGCTCATGGCGA-3') produjo un amplicón esperado de 165 nt, cuya secuencia fue confirmada por secuenciación de Sanger (100% de identidad de nt con la secuencia HTS).
Análisis posteriores sobre 64 árboles mostraron que 17 de ellos estaban infectados por PeSV. PeSV no se detectó en plantas asintomáticas. La mayor parte de árboles sintomáticos dieron positivo para PeSV aunque en alguno de ellos no se detectó por el método desarrollado lo que pone en evidencia la necesidad de mejorar los métodos de detección de este virus emergente en un cultivo de importancia en España como es el granado.

Para la primera descripción del granado como hospedado de PeSV y visualización con más detalle de la sintomatología producida por este virus se puede consultar la comunicación del 25 de noviembre de 2020.
Primeras detecciones de plum bark necrosis stem pitting associated virus en cerezo dulce en España (Autora: A.B. Ruiz-García)
Se realizó un análisis adicional mediante el mapeo de las lecturas contra todas las secuencias completas de PBNSPaV disponibles en las bases de datos utilizando el software Geneious. Los mejores resultados de mapeo se obtuvieron usando el aislado Pair-2 (KC590345) de Francia como referencia, lo que permitió la recuperación de una secuencia de 14.199 nt, que representa el genoma casi completo del aislado P7 (número de acceso de GenBank MN228561).
La identidad de nucleótidos entre el aislado P7 y el Par-2 fue del 99,27 %. Para confirmar la presencia de PBNSPaV en la muestra P7, se realizó la amplificación de una región parcial del gen CP por RT-PCR utilizando los cebadores específicos PBN-CP-F y PBN-CP-R. El producto de PCR de 301 pb obtenido (MN240523) fue secuenciado por Sanger y confirmó con 100% de identidad de la secuencia P7 recuperada por HTS (excluyendo los cebadores usados para la amplificación).
Un total de 24 muestras recogidas de la misma zona de cultivo de cerezo se analizaron mediante RT-PCR utilizando los mismos cebadores. Siete de estas muestras dieron positivo para PBNSPaV, lo que confirma aún más la presencia del virus en cerezp dulce en España. Este es el primer informe de PBNSPaV infectando cerezo dulce en España, lo que contribuye a una mejor comprensión de la epidemiología y la distribución del rango de huéspedes de este patógeno.
Actualización sobre las especies de virus que infectan vid (Autora: A.B. Ruiz-García)
A continuación una actualización de los virus que infectan vid:
Recordando que el virus de los cítricos Citrus tatter feaf ha sido detectado por primera vez en Europa (Autora: A.B. Ruiz-García)
Actualmente la ICTV lo considera como una cepa de un virus conocido de frutales, el Apple stem grooving virus (ASGV).
La mayoría de las variedades de cítricos comerciales infectadas son asintomáticas excepto cuando se propagan sobre patrones Poncirus trifoliata o Poncirus trifoliata x Citrus sinensis. Es entonces cuando los árboles presentan enanismo, hojas cloróticas, incompatibilidad en la unión del injerto lo que conlleva a al deterioro final del árbol. Esto supone un problema grave porque los patrones basados en Poncirus trifoliata y sus híbridos se utilizan ampliamente en todas las zonas productoras del mundo por su tolerancia al virus de la tristeza de los cítricos y a las especies de Phytophthora.
Este virus se describió por primera vez en California y posteriormente en Australia, Korea, Nigeria, Japan, Sudafrica, y China.
Sin embargo en el año 2019 (Turgut Alas et al., Saudi Journal of Biological Sciences 26, 995-998) detectaron este virus Chipre, siendo la primera detección del este virus en cítricos en Europa.
La sintomatología que los autores observaron fue la siguiente:

Distorsión de hojas, necrosis y síntomas cloróticos en hojas infectadas con CTLV (T. Alas et al. 2019)
Para la identificación del virus emplearon RT-PCR convencional empleando los iniciadores específicos:
TL1F: TGAAAACCTTTGCTGCCACCTCT
TL1R: TACTCTCCGAACCTGCCTCGAAA
que amplificaba un fragmento de 309 pb a una temperatura de annealing de 59ºC
Se trata pues de una detección muy a tener en cuenta puesto que este virus se ha considerado hasta la fecha como de cuarentena para la citricultura española.
¿Qué son los aislados Resistance-Breaking (RB) del virus de la tristeza de los cítricos? (Autora: A.B. Ruiz-García)
Tener controlada mediante patrones tolerantes la enfermedad de la tristeza de los cítricos causada por el virus CTV y que ha producido la muerte de más de 50 millones de árboles sólo en España, no quiere decir que debamos olvidar esta problemática.
Los virus evolucionan, se adaptan, mutan y recombinan para vencer resistencias de hospedadores y avanzar en su objetivo, su supervivencia. Y así han aparecido y se han descrito nuevos grupos o genotipos de CTV, como el caso de los aislados Resistance Breaking (RB) formados por un grupo de aislados con características genéticas y biológicas particulares, que se han dividido en dos genotipos, los del grupo 1 RB y los del grupo 2 RB.
Los aislados RB, a diferencia del resto de aislados del virus de la tristeza, pueden replicarse y moverse de forma sistémica en Poncirus trifoliata, que muestra resistencia a otros aislados de CTV.
Estos genotipos RB, se han descrito en Nueva Zelanda, República Dominicana, Puerto Rico, Sudáfrica, California, Brasil y Marruecos.
En España solo existen aislados poco agresivos de CTV, que producen tristeza cuando naranjos dulces, mandarinos, limas y pomelos se injertan sobre naranjo amargo, y se produce una incompatibilidad entre patrón y variedad, por lo que se mantiene como prioridad evitar la entrada de CTV no europeos que incluyen ahora los aislados RB para preservar nuestra citricultura.
Se han diseñado y validado los iniciadores específicos siguientes que permiten realizar PCRs convencionales. El protocolo de detección es el siguiente:
Muestreo: 5 brotes terminales (10 cm) alrededor de la copa del árbol
Purificación de ácidos nucleicos: RNeasy Plant kit o similar
Preparación del cóctel:
Para cada reacción de 25 microlitros (3 microlitros de RNA purificado)
H2O 10,7 microlitros
Tampón 5x Promega 5 microlitros
Cl2Mg 25 mM (uso 1,5 mM) 1,5 microlitros
dNTP (2,5 mM cada uno) uso 0,25 mM 2,5 microlitros
Iniciador F1 (25 microM) uso 1microM 1 microlitro
Inciador R1 (25 microM) uso 1microM 1 microlitro
Promega AMV (10U/microlitro) 0,1 microlitros
GoTaq G2 Promega (5 U/microlitro) 0,2 microlitros
Según el genotipo se seleccionan los iniciadores:
RB group1
Iniciador F1 AGT GGT GGA GAT TAC GTT G
Inciador R1 TAC ACG CGA CAA ATC GAG
RB group 2
Iniciador F1 CGG AAG GGA CTA CGT GGT
Iniciador R1 CGT TTG CAC GGG TTC AAT G
Condiciones del Termociclador
40 min -----------------42ºC
4 min ------------------- 94ºC
40 ciclos:
30 s ---------------------- 92ºC
30 s ---------------------- 60ºC
1 min -------------------- 72ºC
10 min ----------------- 72ºC
El granado nuevo hesped del virus passiflora edulis symptomless virus (Autora: A.B. Ruiz-García)

Figura 1: Síntomas producidos por la infección causada por PeSV en granado. Anillos cloróticos, clorosis nervial y anillos cloróticos a lo largo de las nervaduras centrales.
La estructura genómica de diversos aislados del virus se han obtenido y es común la confección de diferentes variantes virales en el mismo árbol.

Figura 2. Genoma del aislado (PeSV)-PS1-5.
Además en este estudio, se realizó microscopio electrónica y se observaron partículas similares a las de los potyvirus. Se emplearon plantas de granado sintomáticas para extraer los viriones. Los viriones que se observaron eran filamentos flexibles y con una longitud de entre 760 a 780 nm y de 11 a 20 nm de ancho, con simetría helicoidal.
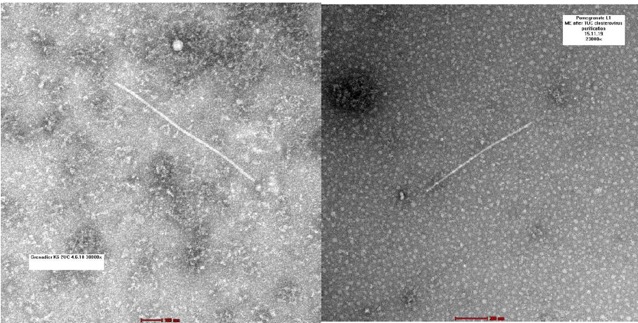
Figura 3. Microscopía electrónica empleando un microscopio de transmisión. La escala corresponde a 100nm en la fotografía de la izquierda y a 200 nm en la de la derecha.
Apple stem pitting virus, un virus de amplia distribución y que infecta diversas especies vegetales (Autora: A.B. Ruiz-García)
En un estudio realizado en nuestro laboratorio en una prospección de níspero en la región de Segorbe, hemos detectado unas variantes capaces de infectar el níspero aunque sin producir sintomatología.
Para avanzar en el conocimiento y poder disponer de herramientas de diagnóstico de estas variantes de níspero, se ha secuenciado completamente un aislado y caracterizamos parcialmente 10 aislados más para ver filogenéticamente donde se situaban.
La estructura del aislado de níspero muestra que mantiene los cinco ORFs (RdRP, TGB1, TGB2, TGB3 y CP) como se muestra en la figura
El análisis filogenético del aislado completo de ASPV lo sitúa próximo a las variantes de peral en color verde.
El níspero se confirma como nuevo huésped de ASPV y se han diseñado iniciadores adaptados a la secuencia del aislado del níspero que ha permitido evaluar la prevalencia en Segorbe que ha resultado ser del 15%.
Un misterio resuelto, el virus asociado al amarilleamiento de la hoja del olivo aporta datos clave para la creación de un nuevo género en la familia Closteroviridae, para el que se propone el nombre Olivavirus (Autora: A.B. Ruiz-García)
El virus asociado al amarilleamiento de la hoja del olivo (OYLaV) se describió por primera vez en 1999, en Italia, en olivos de la variedad Biancolilla. Estos olivos mostraban decoloraciones del verde en las hojas, y tenían una coloración amarillo brillante. Sin embargo sólo se logró una caracterización parcial de un gen (HSP70) pero esta pequeña secuencia permitió que pudiera ser asignado a la familia Closteroviridae.
Estudios posteriores sólo avanzaron parcialmente en su conocimiento del genoma, ya que únicamente se logró conocer alrededor de 5000 nucleótidos. Con este conocimiento se diseñaron técnicas de diagnóstico y se averiguó que OLYaV era uno de los virus del olivo más extendidos en muchas zonas donde se cultivaba. Así, se descubrieron diferentes niveles de incidencia: California (EE. UU.) (93%), Italia (64-21%), Túnez (49%), Líbano (24%), Siria (15%), Grecia (5%) y Albania (2%).
Sin embargo OLYaV permanecía como una especie no asignada en la familia Closteroviridae porque se necesitaban más datos biológicos y moleculares para su clasificación.
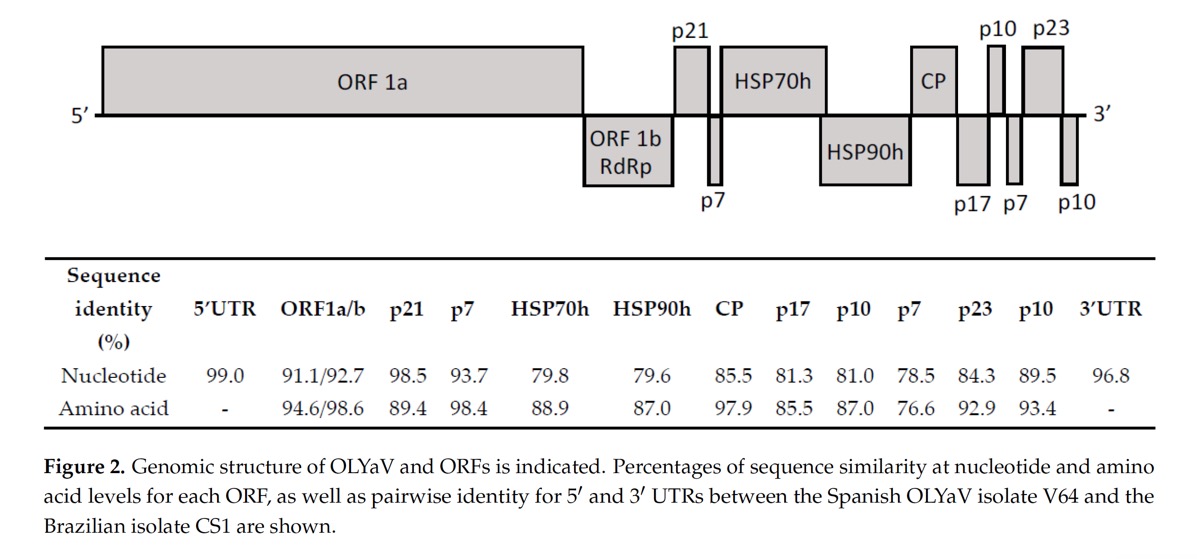
En un estudio que recientemente hemos publicado, hemos descubierto la secuencia completa de OYLaV de 16700 nucleótidos, incluyendo todo su potencial de codificación.
En la figura se muestra la organización genómica de OLYaV. L-Pro: proteasa leader papain-like; Met-T: dominio metal transferasa; Helicase: dominio de la helicasa viral; RdRp: polimerasa RNA dependiente; Las flechas azules indican la posición de corte del proteasa.
La secuencia completa muestra un genoma con estructura típica de la familia Closteroviridae. Contiene 11 pautas de lectura abiertas (ORF,) entre ellos el complejo ORF1ab que codifica para los tres dominios conservados típicos, proteasa líder similar a la papaína, metiltransferasa y helicasa y además la RdRp, además de los genes de las proteínas HSP70 y HSP90.
OLYaV exhibe dos características genómicas originales. Primero, la ORF2 codifica una proteína similar a la taumatina y segundo, no codifica una proteína CPm. Curiosamente, estas dos características genómicas son compartidas por otras dos especies de Closteroviridae no asignadas, PVB and AV1. Además el análisis filogenético, fundamentalmente el estudio de los genes comunes en todas las especies de closterovirus (ORF1a, ORF1b (RdRp), HSP70, HSP90 y CP) ha abierto nuevas posibilidades en la clasificación taxonómica de OLYaV y del persimmon virus B (PVB) y el actidinia virus 1 (AV1), lo que sugiere de forma consistente que forman un nuevo género dentro de la familia Closteroviridae.
Primera detección del Grapevine asteroid mosaic associated virus en viñas de Tempranillo y Macabeo en España (Autora: A.B. Ruiz-García)
Además de estos se descubrió la presencia del virus asteroide de la vid y la secuencia completa se recuperó siendo de 6692 nucletótidos. El análisis filogenético mostró que el aislado español se situaba en un clado diferente a los que previamente estaban descritos, como se muestra en la figura.
Basándonos en la secuencia del aislado TS1 diseñamos dos iniciadores - GAMaV-6010F, 5’CCCTCCTCCTAGCGACGACC3’ y GAMaV- 6426R, 5’GGGTTGAGACGGCGGAGATC3’ - que amplifican un fragmento de 417 nt en la región de la cáspida y que ha permitido detectar los aislados españoles con fiabilidad, revelando una incidencia en el area estudiada de casi el 10%, puesto que los análisis con los iniciadores descritos por otros autores no detectaban los españoles. Estas han sido las primeras detecciones en España de este virus.
Clasificación de virus vegetales de doble cadena de RNA (Autora: A.B. Ruiz-García)
Familia Amalgaviridae
- Género
- Amalgavirus
Familia Partitiviridae
- Géneros
- Alphapartitivirus
- Betapartitivirus
- Deltapartitivirus
Familia Reoviridae
Subfamilia Spinareovirinae
- Géneros
- Fijivirus
- Oryzavirus
Subfamilia Sedoreovirinae
- Género
- Phytoreovirus
Muestreo para el virus de la sharka (Autora: A.B. Ruiz-García)
Se han de tomar si es posible muestras con síntomas típicos: flores, hojas y frutos que presenten los síntomas
En caso de ser muestras sin síntomas: brotes de por lo menos un año con hojas maduras o completamente desarrolladas de la parte central de cada una de las ramas principales (la detección no es fiable en brotes de menos de un año). Las muestras deberían tomarse, como mínimo, en cuatro sitios diferentes (p. ej., cuatro ramas o cuatro hojas) de cada planta; esto es indispensable ya que la distribución del PPV es desigual
El muestreo no debería realizarse durante los meses de temperaturas más altas
Las pruebas son menos fiables si se realizan con muestras tomadas durante el otoño que con las obtenidas al principio de la primavera
En primavera, las muestras pueden ser flores, brotes con las hojas totalmente desarrolladas o frutos.
En verano y en otoño pueden utilizarse para el análisis las hojas maduras y la piel de los frutos maduros recogidos del campo o de los lugares de embalaje.
Las flores, las hojas, los brotes y la piel del fruto pueden almacenarse a una temperatura de 4ºC por no más de 10 días antes del procesamiento.
Los frutos pueden almacenarse durante un mes a una temperatura de 4ºC antes del procesamiento.
En invierno pueden seleccionarse las yemas dormidas o los tejidos de corteza de la zona basal de ramillas, brotes y ramas, o espolones enteros.
La psorosis de los cítricos severa transmisible B (Autora: A.B. Ruiz-García)
La psoriasis severa transmisible o Psoriasis B se encuentra en Argentina y en Uruguay y está ausente en la región EPPO.
La transmisión se ha asociado por un lado al hongo Olpidoium brassiacae
Síntomas
Presencia de descamaciones en la corteza del tronco y ramas
Los síntomas se inician cuando el árbol tiene de 10-15 años con pequeñas lesiones en la corteza
Las lesiones se levantan, se secan y se van desprendiendo
La psorosis B causa descamamiento exuberante en ramas delgadas de los árboles incluso en ramas de menos de 1cm de diámetro
Otras características son que algunas lesiones exudan goma en los límites de las zonas descamadas y en corte transversal de ramas afectadas en la madera se observan zonas de color pardo y de forma irregular que son exudaciones de goma que invaden los vasos de xilema.
Primeras infecciones mixtas en cerezo por diferentes genotipos del virus de la cereza pequeña (Autora: A.B. Ruiz-García)


Este tipo de infecciones mixtas en cerezo con diferentes genotipos de LChV-1 no habían sido descritas hasta la fecha. Ya que no existe vector conocido del virus, la coexistencia de estas variantes podría ser atribuida a prácticas agronómicas como el injerto. Estas infecciones mixtas pueden tener implicaciones en la patogenicidad del virus ya que pueden además producirse recombinaciones. La presencia de dos genotipos diferentes además sugiere dos eventos diferentes de introducción en España.
En un estudio que hemos realizado en colaboración con investigadores de Francia, Grecia y Eslovaquia, hemos logrado un avance en el conocimiento de la diversidad del virus ampliando las cuatro agrupaciones o “clusters” que se conocían a cinco. Estos avances permiten el diseño de nuevas herramientas de diagnóstico capaces de detectar todas las variantes de LChV-1 conocidas hasta el momento.
En la figura de abajo se muestra un árbol filogenético (Maximum likelihood; 500 réplicas) que relaciona las secuencias completas disponibles hasta la fecha del LChV-1.
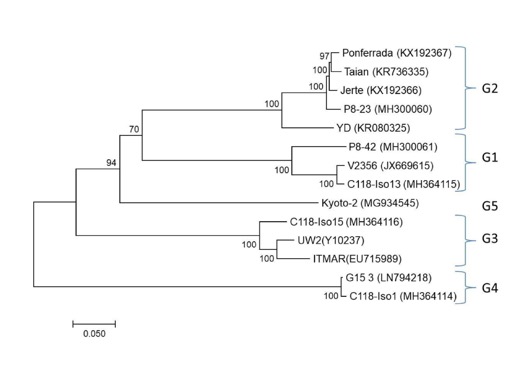
De vuelta con el Grapevine pinot gris virus, y los que no disponen de PCR a tiempo real? (Autora: A.B. Ruiz-García)
Pues con una RT-PCR convencional y para ello os indicamos unos iniciadores que amplifican diferentes regiones del genoma del virus.
Nuestra recomendación, al menos utilizar dos PCRs diferentes, porque este virus es variable y así disminuimos posibilidades de falsos negativos.
Iniciadores de Beuve et al.:
Proteína de movimiento (770pb)
Pg-Mer-F1 5′-GGAGTTGCCTTCGTTTACGA-3′
Pg-Mer-R1 5′-GTACTTGATTCGCCTC GCTCA-3′
Iniciadores de Glasa et al.:
5’UTR (618pb)
GPG-14F 5’-AATTGATCCCGTGTAGTGC-3’
GPG-632R 5’-TCCGAGGACGATGAACCTC-3’
Proteína de movimiento (302pb)
GPG-5637F 5’-ATTGCGGAGTTGCCTTCAAG-3’
GPG-5939R 5’-CTGAGAAGCATTGTCCCATC-3’
Proteína de cápside (411pb)
GPG-6609F 5’-GAGATCAACAGTCAGGAGAG-3’
GPG-7020R 5’-GACTTCTGGTGCCTTATCAC-3’
El protocolo para su detección por RT-PCR convencional es el siguiente:
Muestreo: 4 brotes terminales (10 cm) alrededor de la cepa
Purificación de ácidos nucleicos: RNeasy Plant kit o similar
Preparación del cóctel: Para cada reacción de 25 microlitros (3 microlitros de RNA purificado)
H2O 10,7 microlitros
Tampón 5x Promega 5 microlitros
Cl2Mg 25 mM (uso 1,5 mM) 1,5 microlitros
dNTP (2,5 mM cada uno) uso 0,25 mM 2,5 microlitros
Iniciador 1 (25 microM) uso 1microM 1 microlitro
Iniciador 2 (25 microM) uso 1microM 1 microlitros
GoTaq G2 Promega (5 U/microlitro) 0,2 microlitros
AMV Promega (10 U/microlitro) 0,1 microlitro
Condiciones del Termociclador
30 min ------------------ 45ºC
4 min ------------------- 94ºC
40 ciclos: 30 s ---------------------- 92ºC
30 s ---------------------- 50ºC
1 min -------------------- 72ºC
10 min ----------------- 72ºC
Un virus emergente en vid, Grapevine pinot gris virus (Autora: A.B. Ruiz-García)
Desde su primera descripción en Italia, el virus se ha descrito en otros países como Grecia, República Checa, Eslovaquia, Eslovenia, Francia, Alemania, Portugal, Corea del Sur, China, Canadá, EEUU, Turquía, Brasil, Croacia, Rumanía, Ucrania o Australia.
En España nuestro grupo de investigación describió por primera su presencia en el país en el año 2017, infectando variedades de Garnacha, Bobal y Tempranillo. Con el objetivo de comprender y mejorar la epidemiología de la enfermedad y desarrollar estrategias de control hemos desarrollado una RT-PCR a tiempo real que permite la detección y cuantificación del virus, con un limite de 70 copias del virus en material vegetal. También se ha aplicado a la detección en un posible vector Colomerus vitis.

Además hemos descubierto un nuevo polimorfismo en el genoma del virus que hace que la proteína de movimiento sea más pequeña.
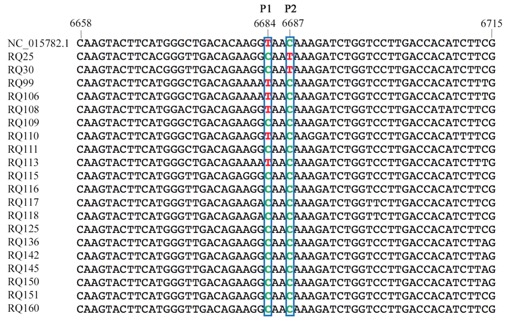
Un estudio filogenético basado en esta región genómica muestra una alta variaibilidad entre los aislados españoles, que se localizan en diversos grupos o “clusters” independientemente de tener o no polimorfismos en la proteína de movimiento (nuevo polimorfismo, círculo rojo completo; polimorfismo previamente descrito por un grupo italiano, círculo con sólo la circunferencia roja)
El desarrollo de esta metodología de detección y el avance en la diversidad del virus, permitirá mejorar herramientas de control y diagnóstico del virus.
La importancia de los insectos vectores en la dipersión de los virus (Autora: A.B. Ruiz-García)
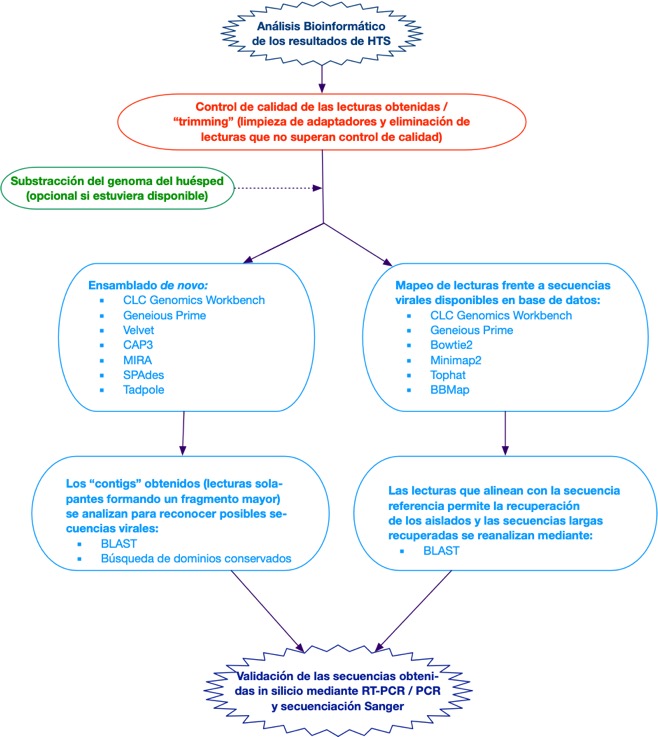
¿Cuál es el esquema del análisis bioinformático de los resultados de secuenciación masiva para el estudio de virus? (Autora: A.B. Ruiz-García)
Los viroides ¿qué son? (Autora: A.B. Ruiz-García)
Su genoma es una molécula de RNA monocatenario circular de 270 a 470 nucleótidos, que no está encapsidada y no es codificante. No tienen pues proteínas propias y son parásitos obligados de las células de la planta huésped.
Se replican de forma autónoma utilizando el sistema de transcripción de las células infectadas. Siguen el modelo de realización del círculo rodante y está relacionado con la naturaleza circular de los viroides.
Hay dos familias de viroides:
Pospiviroidae que se replican en el núcleo
Avsunviroidae que se replican en los cloroplastos
Los viroides invaden la planta huésped por una herida inicial y movimiento célula a célula via plasmodesmos e invasión a larga distancia por los tejidos conductores del floema
La transmisión planta a planta es:
- principalmente mecánica
- pocos casos por pulgones, cuando una molécula de viroide se encapsida en partículas de virus en confección virus-viroide
- por multiplicación vegetativa
- semilla
Los síntomas son similares a los virus fitopatógenos, raquitismo, epinastia, decoloraciones foliares, aclarado de nervios, necrosis, distorsión de hojas, lesiones necróticas locales o generalizadas
El primer viroide descubierto fue el causante de la enfermedad del tubérculo fusiforme de la patata Potato splindle tuber viroid o PSTVd
El segundo viroide fue el de la exocortis de los cítricos, citrus exocortis viroid o CEVd
otro viroide de importancia es el Peach latente mosaic viroid o PLMVd que tiene una sintomatología muy característica en los frutos
Clasificación de virus vegetales de DNA de cadena simple (Autora: A.B. Ruiz-García)
Los más representativos se clasifican en los siguientes géneros
Familia Geminiviridae
Géneros:
* Becurtovirus
* Begomovirus
* Capulavirus
* Curtovirus
* Grablovirus
* Eragrovirus
* Mastrevirus
* Topocuvirus
* Turncurtovirus
Familia Nanoviridae
Géneros:
* Babuvirus
* Nanovirus
¿Cómo evitar la introducción de cepas agresivas tipo M de sharka? (Autora: A.B. Ruiz-García)
temprana de melocotonero en amplias zonas de un país como España que
produce más de 700.000 toneladas de melocotones.
Hay unas medidas importantes que deben conocerse y aplicarse para evitar o reducir los riesgos de
introducción de tipos agresivos del virus de la sharka:
- La más básica: No introducir material vegetal de zonas donde exista PPV M, evitando la toma de varetas de material vegetal especialmente si son recolectadas en el sur de Francia, Italia, Grecia o cualquier país de Europa del Este.
- Una medida lógica: No plantar material vegetal de frutales de hueso sin un análisis previo de PPV y que las plantaciones se realicen con material certificado libre de PPV
- Una medida de atención: Vigilar los árboles en floración, durante el engrosamiento del fruto y en el momento de la recolección y observar si aparecieran síntomas
- Una medida más: Analizar los melocotoneros que presenten síntomas de PPV
¿Por qué no es suficiente con la sensibilidad y la especificidad en el diagnóstico? (Autora: A.B. Ruiz-García)
Fijémonos en la tabla de contingencia de variables de diagnóstico:
La sensibilidad de la prueba diagnóstica se define como la probabilidad de que el resultado de la prueba sea positivo en una planta enferma. Representa la proporción de verdaderos positivos diagnosticados por la técnica entre las plantas enfermas VP/(VP+FN)
La especificidad de la prueba diagnóstica se define como la probabilidad de que el resultado de la prueba sea negativo en una planta sana. Representa el porcentaje de resultados negativos respecto del total de plantas sanas VN/(FP+VN)
La sensibilidad no contempla los falsos positivos y la especificidad no contempla los falsos negativos que rinde la técnica. Y es precisamente esto lo que necesita conocer el técnico o lo que es lo mismo saber ¿cúal es la probabilidad de que un resultado positivo por la técnica sea un verdadero positivo y no un falso positivo, independientemente de su sensibilidad? y ¿cúal es la probabilidad de que un resultado negativo por la técnica sea un verdadero negativo y no un falso negativo, independientemente de su especificidad? La respuesta la dan los valores predictivos positivo y negativo, que se calculan considerando las filas de la tabla de contingencia al contrario que la sensibilidad y especificidad que se calculan en las columnas de la tabla de contingencia.
El valor predictivo positivo de una prueba diagnóstica se define como la probabilidad de que la planta esté enferma habiendo dado positivo en la prueba. Así pues, el valor predictivo positivo representa el porcentaje de plantas realmente enfermas respecto del total de plantas que han dado positivo VP/(VP+FP). Un valor alto indica que la probabilidad de que la planta esté realmente enferma habiendo dado positivo en la prueba diagnóstica es muy alta.
El valor predictivo negativo se define como la probabilidad de que una planta esté sana habiendo dado negativo. Representa el porcentaje de plantas sanas respecto del total de plantas que han dado negativo en la prueba VN/(VN+FN).
Sin embargo, si aplicamos el teorema de Bayes se puede concluir que los valores predictivos dependen de la prevalencia de la enfermedad que tratamos de diagnosticar según la siguiente relación: valor predictivo positivo= (sensibilidad de la prueba x prevalencia de la enfermedad) / ((sensibilidad x prevalencia) + [(1 - especificidad) x (1 - prevalencia)]). Como puede observarse, cuanto mayor sea la prevalencia de la enfermedad en la población mayor será el valor predictivo positivo de la prueba diagnóstica, aún manteniéndose constantes su sensibilidad y especificidad; de forma similar ocurre con el valor productivo negativo.
Así pues, si sensibilidad y especificidad no dan una respuesta apropiada por no contemplar falsos positivos y falsos negativos respectivamente y los valores predictivos positivo y negativo varían con la prevalencia ¿qué parámetros son los apropiados para evaluar la técnica diagnóstica? La respuesta la dan “likelihood ratios” de positivo y negativo o razón de verosimilitud. Así la razón de verosimilitud positiva es la proporción de verdaderos positivos correctamente identificados por la técnica, o sea, la sensibilidad dividido por la proporción de falsos positivos que la técnica diagnostica FP/VP+FP, o lo que es lo mismo 1-especificidad = 1-(VN/(VN+FP)).
La razón de verosimilitud negativa es la proporción de falsos negativos que la técnica diagnostica (1-sensibilidad), dividido por la proporción de verdaderos negativos correctamente identificados por la técnica o lo que es lo mismo, la especificidad
La enorme ventaja de emplear las razones de verosimilitud es que pueden usarse para cuantificar la probabilidad de la enfermedad para una planta individual. El teorema de Bayes se usa para traducir la información suministrada por los las razones de verosimilitud y probabilidad de enfermedad. El teorema de Bayes establece que la razón de probabilidades o razón de momios pre-test, que es lo mismo el cociente de la probabilidad de enfermedad y la probabilidad de no enfermedad antes de realizar el test, multiplicado por la razón de verosimilitud da lugar a la razón de probabilidades post-test de la enfermedad o sea el cociente de la probabilidad de la enfermedad y la probabilidad de no enfermedad después de realizar el análisis. La probabilidad post-test se puede calcular de la siguiente forma:
Probabilidad Pre-test = Prevalencia
Razón de probabilidades Pre-test = Prevalencia/(1-Prevalencia)
Razón de probabilidades Post-test = Razón de probabilidades Pre-test x Razón de verosimilitud
Probabilidad Post-test = Razón de probabilidades Post-test /(1+ Razón de probabilidades Post-test)
Además las razones de verosimilitud de diversos métodos pueden combinarse en la formula de modo que: Razón de probabilidades Post-test = Razón de probabilidades Pre-test x Razón de verosimilitud 1 x Razón de verosimilitud 2 x Razón de verosmilitud 3.
Así pues la probabilidad pre-test de enfermedad puede compararse con la probabilidad post-test. La diferencia entre la previa y la posterior es una manera muy efectiva de analizar la eficiencia de un método diagnóstico.
El enanismo de la satsuma (Autora: A.B. Ruiz-García)
El virus se transmite rápido por injerto entre cítricos y mecánicamente entre cítricos y huéspedes no cítricos. Su dispersión natural parece ser que es por vectores del suelo en las comarcas de Japón y también se ha indicado una posible transmisión entre Viburnum odoratissimum y cítricos.
La enfermedad produce raquitismo y las hojas se abarquillan y adquieren forma de cuchara.
Los frutos son de baja calidad y bajo valor comercial y es una amenaza para los países productores.
El enanismo de la satsuma severa es un problema en Japón.
Amenazas del exterior II: otro virus de vid peligroso (Autora: A.B. Ruiz-García)
Es importante conocer cómo poder detectarlo en material vegetal por PCR, por lo que abajo hay un protocolo de PCR para su diagnóstico.
Muestreo: 4 brotes terminales (10 cm) alrededor de la cepa
Purificación de ácidos nucleicos: DNeasy Plant kit o similar
Preparación del cóctel: Para cada reacción de 25 microlitros (3 microlitros de DNA purificado)
H2O 10,8 microlitros
Tampón 5x Promega 5 microlitros
Cl2Mg 25 mM (uso 1,5 mM) 1,5 microlitros
dNTP (2,5 mM cada uno) uso 0,25 mM 2,5 microlitros
GVCV-F1 (25 microM) uso 1microM 1 microlitro
GVCV-R1 (25 microM) uso 1microM 1 microlitro
GoTaq G2 Promega (5 U/microlitros) 0,2 microlitros
ORF1 (557pb)
GVGF1 5′-CTCGTCGCATTTGTAAGA-3′ (Al Rwahnih et al.)
GVGR1 5′-ACTGACAAGGCCTACTACG-3′ (Al Rwahnih et al.)
Condiciones del Termociclador
4 min ------------------- 94ºC
40 ciclos: 30 s ---------------------- 92ºC
30 s ---------------------- 50ºC
1 min -------------------- 72ºC
10 min ----------------- 72ºC
Amenazas del exterior I: un virus de vid peligroso (Autora: A.B. Ruiz-García)
Entre ellos está un Badnavirus, un virus de DNA, que se denomina grapevine vein clearing virus. Ha sido el primer virus de DNA descrito en vid (familia Caulimoviridae, género Badnavirus), que produce el síndrome de decaimiento y aclaramiento de las nervaduras y que ha sido descrito en los estados de Missouri, Illinois e Indiana (EEUU).
El protocolo para su detección por PCR convencional es el siguiente:
Muestreo: 4 brotes terminales (10 cm) alrededor de la cepa
Purificación de ácidos nucleicos: DNeasy Plant kit o similar
Preparación del cóctel: Para cada reacción de 25 microlitros (3 microlitros de DNA purificado)
H2O 10,8 microlitros
Tampón 5x Promega 5 microlitros
Cl2Mg 25 mM (uso 1,5 mM) 1,5 microlitros
dNTP (2,5 mM cada uno) uso 0,25 mM 2,5 microlitros
GVCV-F1 (25 microM) uso 1microM 1 microlitro
GVCV-R1 (25 microM) uso 1microM 1 microlitros
GoTaq G2 Promega (5 U/microlitro) 0,2 microlitros
ORF3 (530pb)
GVCV-F1 5′-CACGTTTCAAAGAAAGATGGAC-3′ (Zhang et al.)
GVCV-R1 5′-ATCCKTCCATGCAWCCGTCAG-3′ (Zhang et al.)
Condiciones del Termociclador
4 min ------------------- 94ºC
40 ciclos: 30 s ---------------------- 92ºC
30 s ---------------------- 50ºC
1 min -------------------- 72ºC
10 min ----------------- 72ºC
Metodología bayesiana con modelos latentes (Autora: A.B. Ruiz-García)
A continuación se detalla la programación del modelo:
Código de OpenBUGS, que asume dos técnicas condicionalmente dependientes DAS-ELISA (ELISA) y spot real-time RT-PCR (spot), y una tercera independiente, real-time RT-PCR convencional (qPCR), y tres poblaciones. Priors informativos para sensibilidad (Se) y especificidad (Sp) de DAS-ELISA y real-time RT-PCR convencional. Priors no informativos, β (1,1), para sensibilidad y especificidad de spot real-time RT-PCR y prevalencias de tres poblaciones (Prev1, Prev2, Prev3). Likelihood ratios (LR) se calculan en cada iteración.
model;
{
y1[1:Q, 1:Q, 1:Q] ~ dmulti(p1[1:Q, 1:Q, 1:Q], n1)
y2[1:Q, 1:Q, 1:Q] ~ dmulti(p2[1:Q, 1:Q, 1:Q], n2)
y3[1:Q, 1:Q, 1:Q] ~ dmulti(p3[1:Q, 1:Q, 1:Q], n3)
p1[1,1,1] <- Prev1*SeqPCR*(Sespot*SeELISA+covDp) + (1-Prev1)*(1-SpqPCR)*((1-Spspot)*(1-SpELISA)+covDn)
p1[1,2,1] <- Prev1*SeqPCR*(Sespot*(1-SeELISA)-covDp) + (1-Prev1)*(1-SpqPCR)*((1-Spspot)*SpELISA-covDn)
p1[1,1,2] <- Prev1*(1-SeqPCR)*(Sespot*SeELISA+covDp) + (1-Prev1)*SpqPCR*((1-Spspot)*(1-SpELISA)+covDn)
p1[1,2,2] <- Prev1*(1-SeqPCR)*(Sespot*(1-SeELISA)-covDp) + (1-Prev1)*SpqPCR*((1-Spspot)*SpELISA-covDn)
p1[2,1,1] <- Prev1*SeqPCR*((1-Sespot)*SeELISA-covDp) + (1-Prev1)*(1-SpqPCR)*(Spspot*(1-SpELISA)-covDn)
p1[2,2,1] <- Prev1*SeqPCR*((1-Sespot)*(1-SeELISA)+covDp) + (1-Prev1)*(1-SpqPCR)*(Spspot*SpELISA+covDn)
p1[2,1,2] <- Prev1*(1-SeqPCR)*((1-Sespot)*SeELISA-covDp) + (1-Prev1)*SpqPCR*(Spspot*(1-SpELISA)-covDn)
p1[2,2,2] <- Prev1*(1-SeqPCR)*((1-Sespot)*(1-SeELISA)+covDp) + (1-Prev1)*SpqPCR*(Spspot*SpELISA+covDn)
p2[1,1,1] <- Prev2*SeqPCR*(Sespot*SeELISA+covDp) + (1-Prev2)*(1-SpqPCR)*((1-Spspot)*(1-SpELISA)+covDn)
p2[1,2,1] <- Prev2*SeqPCR*(Sespot*(1-SeELISA)-covDp) + (1-Prev2)*(1-SpqPCR)*((1-Spspot)*SpELISA-covDn)
p2[1,1,2] <- Prev2*(1-SeqPCR)*(Sespot*SeELISA+covDp) + (1-Prev2)*SpqPCR*((1-Spspot)*(1-SpELISA)+covDn)
p2[1,2,2] <- Prev2*(1-SeqPCR)*(Sespot*(1-SeELISA)-covDp) + (1-Prev2)*SpqPCR*((1-Spspot)*SpELISA-covDn)
p2[2,1,1] <- Prev2*SeqPCR*((1-Sespot)*SeELISA-covDp) + (1-Prev2)*(1-SpqPCR)*(Spspot*(1-SpELISA)-covDn)
p2[2,2,1] <- Prev2*SeqPCR*((1-Sespot)*(1-SeELISA)+covDp) + (1-Prev2)*(1-SpqPCR)*(Spspot*SpELISA+covDn)
p2[2,1,2] <- Prev2*(1-SeqPCR)*((1-Sespot)*SeELISA-covDp) + (1-Prev2)*SpqPCR*(Spspot*(1-SpELISA)-covDn)
p2[2,2,2] <- Prev2*(1-SeqPCR)*((1-Sespot)*(1-SeELISA)+covDp) + (1-Prev2)*SpqPCR*(Spspot*SpELISA+covDn)
p3[1,1,1] <- Prev3*SeqPCR*(Sespot*SeELISA+covDp) + (1-Prev3)*(1-SpqPCR)*((1-Spspot)*(1-SpELISA)+covDn)
p3[1,2,1] <- Prev3*SeqPCR*(Sespot*(1-SeELISA)-covDp) + (1-Prev3)*(1-SpqPCR)*((1-Spspot)*SpELISA-covDn)
p3[1,1,2] <- Prev3*(1-SeqPCR)*(Sespot*SeELISA+covDp) + (1-Prev3)*SpqPCR*((1-Spspot)*(1-SpELISA)+covDn)
p3[1,2,2] <- Prev3*(1-SeqPCR)*(Sespot*(1-SeELISA)-covDp) + (1-Prev3)*SpqPCR*((1-Spspot)*SpELISA-covDn)
p3[2,1,1] <- Prev3*SeqPCR*((1-Sespot)*SeELISA-covDp) + (1-Prev3)*(1-SpqPCR)*(Spspot*(1-SpELISA)-covDn)
p3[2,2,1] <- Prev3*SeqPCR*((1-Sespot)*(1-SeELISA)+covDp) + (1-Prev3)*(1-SpqPCR)*(Spspot*SpELISA+covDn)
p3[2,1,2] <- Prev3*(1-SeqPCR)*((1-Sespot)*SeELISA-covDp) + (1-Prev3)*SpqPCR*(Spspot*(1-SpELISA)-covDn)
p3[2,2,2] <- Prev3*(1-SeqPCR)*((1-Sespot)*(1-SeELISA)+covDp) + (1-Prev3)*SpqPCR*(Spspot*SpELISA+covDn)
ls <- (Sespot-1)*(1-SeELISA)
us <- min(Sespot,SeELISA) - Sespot*SeELISA
lc <- (Spspot-1)*(1-SpELISA)
uc <- min(Spspot,SpELISA) - Spspot*SpELISA
rhoD <- covDp / sqrt(Sespot*(1-Sespot)*SeELISA*(1-SeELISA))
rhoDc <- covDn / sqrt(Spspot*(1-Spspot)*SpELISA*(1-SpELISA))
covDn ~ dunif(lc, uc)
covDp ~ dunif(ls, us)
SeELISA~dbeta(108.41,18.49)
SpELISA~dbeta(34.17,1.34)
Sespot~dbeta(1,1)
Spspot~dbeta(1,1)
SeqPCR~dbeta(53.58,2.63)
SpqPCR~dbeta(51.41,7.23)
Prev1~dbeta(1,1)
Prev2~dbeta(1,1)
Prev3~dbeta(1,1)
LRELISApos<-SeELISA/(1-SpELISA)
LRELISAneg<-(1-SeELISA)/SpELISA
LRspotpos<-Sespot/(1-Spspot)
LRspotneg<-(1-Sespot)/Spspot
LRqPCRpos<-SeqPCR/(1-SpqPCR)
LRqPCRneg<-(1-SeqPCR)/SpqPCR
LRELISAposspotpos<-LRELISApos*LRspotpos
LRELISAposspotneg<-LRELISApos*LRspotneg
LRELISAnegspotpos<-LRELISAneg*LRspotpos
LRELISAnegspotneg<-LRELISAneg*LRspotneg
LRELISAposqPCRpos<-LRELISApos*LRqPCRpos
LRELISAposqPCRneg<-LRELISApos*LRqPCRneg
LRELISAnegqPCRpos<-LRELISAneg*LRqPCRpos
LRELISAnegqPCRneg<-LRELISAneg*LRqPCRneg
LRspotposqPCRpos<-LRspotpos*LRqPCRpos
LRspotposqPCRneg<-LRspotpos*LRqPCRneg
LRspotnegqPCRpos<-LRspotneg*LRqPCRpos
LRspotnegqPCRneg<-LRspotneg*LRqPCRneg
LRELISAposspotposqPCRpos<-LRELISApos*LRspotpos*LRqPCRpos
LRELISAposspotposqPCRneg<-LRELISApos*LRspotpos*LRqPCRneg
LRELISAposspotnegqPCRpos<-LRELISApos*LRspotneg*LRqPCRpos
LRELISAposspotnegqPCRneg<-LRELISApos*LRspotneg*LRqPCRneg
LRELISAnegspotposqPCRpos<-LRELISAneg*LRspotpos*LRqPCRpos
LRELISAnegspotposqPCRneg<-LRELISAneg*LRspotpos*LRqPCRneg
LRELISAnegspotnegqPCRpos<-LRELISAneg*LRspotneg*LRqPCRpos
LRELISAnegspotnegqPCRneg<-LRELISAneg*LRspotneg*LRqPCRneg
}
#Note: for this model initial values were:
#SeELISA=0.86,SpELISA=0.99,SeqPCR=0.97,SpqPCR=0.89
Con estos parámetros se puede establecer la probabilidad post-test de que una cepa esté infectada según los resultados del diagnóstico de una, dos o tres técnicas. De esta forma dependiendo de la prevalencia de la infección se puede seleccionar el método más apropiado para realizar el análisis.
¿Cómo se interpreta una curva de probabilidad post-test en diagnóstico? (Autora: A.B. Ruiz-García)
La probabilidad pre-test es la prevalencia de la enfermedad en el área de estudio o en el conjunto de muestras analizadas. la linea recta diagonal divide los resultados positivos (arriba de ella) de los negativos (debajo de ella).
Así como se puede observar a baja prevalencia el ELISA+ junto con una PCR+ dan una probabilidad post-test, es decir una seguridad de que la muestra sea positiva muy alta. Comparando un resultado positivo en este modelo por ELISA y por PCR vemos que a baja prevalencia un resultado positivo es más fiable que un resultado positivo por PCR que tiene una probabilidad post-test más bajo. A altas prevalencia los resultados positivos tienden a igualarse en su probabilidad.
¿Pero y que pasa para los resultados negativos? A bajas prevalencias tanto ELISA- como PCR- como ELISA y PCR- tienden a ser similares sus probabilidades post test, luego cualquiera tiene bastante fiabilidad a bajas prevalecías para los resultados negativos. A altas prevalencia es mejor la PCR porque su probabilidad post-test es decir un falso negativo es menor que el ELISA que un negativo es posible que sea un falso negativo.
Con este tipo de curvas es posible aplicar técnicas de diagnóstico con criterio.
El protocolo oficial de la EPPO para la detección del virus de la sharka (Autora: A.B. Ruiz-García)
El enanismo clorótico de los cítricos (Autora: A.B. Ruiz-García)
El rendimiento y producción de los árboles afectados se reduce sustancialmente.
El agente causal el Citrus cholortic dwarf associated virus que tiene como huéspedes, limonero, pomelo, mandarino tangelo y naranjo dulce.
Se transmite por mosca blanca
En campo afecta a todas las variedades de limonero y la mayor parte de mandarinos e híbridos
Sus síntomas son moderados en pomelo y suaves en naranjo dulce lo que puede suponer que al no ser severos sirvan como reservorios del virus y fuente de inóculo.
El tamaño de la planta se reduce las hojas se deforman y hay una fuerte clorosis.
Dispersión de virus a larga distancia (Autora: A.B. Ruiz-García)
La leprosis de los cítricos (Autora: A.B. Ruiz-García)
- Citrus leprosis virus tipo citoplásmico (CiLV-C1 y CiLV-C2)
- Citrus leprosis virus tipo nuclear (CiLV-N)

Huéspedes:
Citrus spp.
Pomelo (C. paradisi)
Naranjo (C. sinensis)
Limonero (C. limon)
Mandarino (C. reticulata)
Distribución geográfica:
USA (Florida)
Argentina
Brasil
Paraguay
Uruguay
Venezuela
Costa Rica
Panamá
Honduras
Guatemala
Bolivia
México
Colombia
Región EPPO: Ausente
El virus es transmitido por ácaros del género Brevipalpus:
- Brevipalpus californicus
- Brevipalpus obovatus
- Brevipalpus phoenicis

Síntomas:
- Produce pérdidas considerables por el manchado y caída prematura del fruto (superior al 50% de producción), debilitamiento de los árboles como consecuencia de los chancros que causa en las ramas y los brotes.
- Forma lesiones cloróticas, a veces con necrosis central que evoluciona a chancros de 5-6 mm de diámetro. En frutos a veces presentan depresiones pero no afectan a gajos
- En brotes y hojas los chancros son planos o ligeramente prominentes y pueden formar manchas concéntricas o presentar impregnación de goma
- Las frutas con lesiones no tienen valor comercial
- Las manchas en hojas y fruta, su caída temprana y muerte de ramas suponen además un peligro para la planta
Impacto económico:
La enfermedad de la leprosis es una de las enfermedades económicamente más importantes que representa millones de dólares de daños en países donde está establecida, y es una amenaza países productores sin la enfermedad (CABI 2014). Por ejemplo, el control de la leprosis representa en Brasil el 24% del coste de producción sobre todo en el control químico del vector. Desde 1990 es la enfermedad más importante que afecta a la industria brasileña y Panamá detectó la leprosis en 1999 y tiene un programa de erradicación muy costoso.
Clasificación de virus vegetales de RNA de polaridad negativa (Autora: A.B. Ruiz-García)
Los más importantes se clasifican en
Orden Mononegavirales
- Familia Rhabdoviridae
- Género Cytorhabdovirus
- Género Dichoravirus
- Género Nucleorhabdovirus
- Género Varicosavirus
Orden Bunuyavirales
- Familia Fimoviridae
- Género Emaravirus
- Familia Tospoviridae
- Género Orthotospovirus
- Familia Phenuivirdae
- Género Tenuivirus
- No asignado a familia
- Género Coguvirus
Sin Orden
- Familia Aspiviridae
- Género Ophiovirus
La enfermedad de la decoloración de la hoja del roditis (Autora: A.B. Ruiz-García)

Se empleó el chip 318 y se seleccionó una muestra de una viña de 20 años con claros síntomas de la enfermedad.
El análisis de la muestra reveló un nuevo virus con la organización genómica de los virus del género Badnavirus
Su genoma incluía 3 marcos de lectura abiretos (ORFs) y un posible 4º (no típico de Badnavirus)
ORFs 1, 2 and 4 codifican para proteínas de 17.1, 15.1 and 16.2 kDa, mientras que el ORF3 codifica para una poliproteína de 211 kDa (con dominios relacionados con movimiento, encapsidación, proteasa, transcriptas reversa y RNase H)
El nuevo badnavirus se asoció a la enfermedad, puesto que se detectó en todas las muestras sintomáticas analizadas (20 de 20) y sólo en dos muestras de las 7 asintomáticas analizadas (2 de 7).
Este nuevo virus se ha detectado posteriormente en otros países como Italia y Turquía y se EPPO lo a incluido en sus alertas.
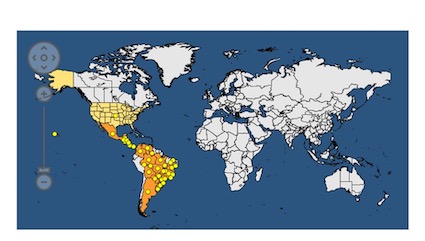
¿Qué es la secuencación masiva? (Autora: A.B. Ruiz-García)
Esto es posible gracias al avance de la tecnología que ha permitido el desarrollo de equipos capaces de realizar de forma simultánea la secuenciación de DNA de millones de fragmentos diferentes de ADN.
Así pues, empleando esta tecnología, es potencialmente posible la detección de cualquier virus que esté presente en una muestra de una planta.

Esta tecnología puede usarse para reemplazar una amplia gama de métodos convencionales, ya que además tiene potencial para identificación de virus tanto conocidos como desconocidos.
Básicamente el proceso es sencillo: 1) se selecciona una muestra; 2) se purifican los ácidos nucleicos de la muestra; 3) se preparan genotecas, es decir los ácidos nucleicos se trocean y se les añaden adaptadores para que sea posible realizar una reacción de PCR a la vez en todas los trozos con los adaptadores; 4) el equipo secuencia millones de estos fragmentos y 5) se realiza un análisis bioinformático para averiguar que virus hay presentes
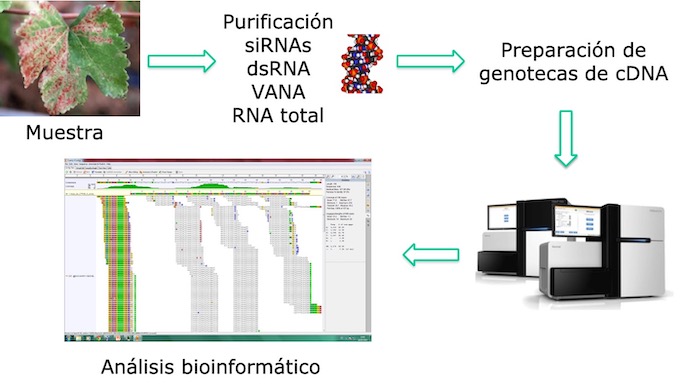
En el campo de la virología vegetal, HTS está revolucionando la comprensión de los científicos sobre la naturaleza ubicua de los virus y demuestra claramente que apenas estamos comenzando a conocer la gran diversidad viral previamente desconocida.
Se puede aplicar para abordar diferentes campos de la virología:
-Etiología viral
-Caracterización de virus
-Taxonomía
-Genética de poblaciones
-Desarrollo de métodos de diagnóstico
En los últimos años se han descrito una gran cantidad de virus nuevos mediante el empleo de esta tecnología, se ha propuesto la creación de nuevos géneros y el conocimiento adquirido ha conllevado una continua revisión de la taxonomía.
Otros virus de cítricos de gran importancia (Autora: A.B. Ruiz-García)
Entre los virus de los cítricos no presentes en la UE destacan:
El virus que se asocia a la enfermedad de la clorosis nervial de los cítricos. Esta enfermedad está causada por un virus recientemente identificado del género Mandarivirus la especie Citrus yellow vein clearing virus.
Este virus se ha encontrado en China, Pakistán, Turquía, India e Irán.
Fundamentalmente afecta a limonero produciendo una gran necrosis y clorosis nervial, además de distorsión de hojas en limonero con reducción significativa de producción y calidad de fruta. Además se transmite por vectores A. spireacola y Dialeurodes citri.
Otro virus importante es el virus del mosaico amarillo de los cítricos presente en India. Es un virus transmitido por Planococcus citri. Produce síntomas en mosaico en hojas infectadas. Produce síntomas en naranjo amargo, reduciendo el tamaño de las hojas y hace que los árboles infectados sean raquíticos. El agente causal es Citrus yellow mosaic virus. Es un Badnavirus que puede integrarse en el genoma de la planta y por tanto su control es muy difícil y es de cuarentena en muchos países incluyendo EEUU o Nueva Zelanda.
La psoriasis severa transmisible, es una enfermedad de importancia en los países que la tienen. Esta enfermedad está producida por Citrus psorosis virus B, un Ophiovirus. La psoriasis tipo B es muy agresiva y tiene un desarrollo rápido, incluso en ramas secundarias. Está persente en Argentina e Uruguay. Se ha sugerido su transmisión por áfidos y se ha asociado a transmisión por el hongo Olpidoium brassiacae.
La muerte súbita de los cítricos de es una enfermedad muy grave que se ha descrito en Brasil, que afecta a árboles injertados sobre lima Rangpur, Citrus volkameriana, Citrus jambiri y Citrus pennivisiculata. El decaimiento del naranjo puede ser tan rápido que el árbol que no se produce la abscisión de los frutos, quedando todavía en el árbol muerto y seco. Se observa una coloración amarilla en el tronco del patrón. La enfermedad se transmite por T. citricida y A. spiraecola. Se ha asociado a la presencia de varios virus entre los que se ha identificado un tymovirus, un genotipo de la especie Citrus sudden death-associated virus.
Otro virus de importancia es el Citrus chlorotic associated dwarf virus. Presente en Turquía. Transmitido por mosca blanca. Afecta a todas las variedades de limonero, a la mayor parte de mandarinos e híbridos, produciendo abolladuras y variaciones en hojas maduras y tamaño reducido de la planta con deformaciones y fuerte clorosis.
Citrus tatter leaf virus es un capillovirus y aunque en la mayoría de cítricos es asintomático el injerto de Poncirus trifoliata con hibridos infectados produce aparición de síntomas graves como enanismo, amarilleamiento, defoliación, mariñaque por encima de la linea de injerto y falta de unión.
Hay dos viroides de cítricos de importancia:
El viroide de la caquexia o xiloporosis Citrus cachexia viroid produce amarilleamiento progresivo de la copa del árbol por pérdida de follaje, detención del crecimiento y finalmente la muerte del árbol. La unión del injerto presenta miriñaque con acanaladuras en tronco impregnadas de goma. De hecho la caquexia es una variante agresiva de Hop stunt viroid.
El viroide de la exocortis Citrus exocortis viroid produce grietas verticales y escamas en la corteza, manchas amarillas y enanismo y decaimiento.
Primera detección de Grapevnie Syrah virus 1 en España (Autora: A.B. Ruiz-García)
El virus de la tristeza de los cítricos en España, un poco de historia (Autora: A.B. Ruiz-García)
En España CTV la muerte de alrededor de 50 millones de árboles entre 1935 y 2009
1935-1989 20 millones / 54 años
1990-2009 30 millones / 19 años
Tras la manifestación epidémica y masiva de 1957 se realizó la primera detección oficial en 1959 y nueve años después en 1968 se prohibió el uso de naranjo agrio como patrón. Entre 1968 y 1975 se importaron variedades extranjeras libres de virus y se realizaron las primeras plantaciones sobre patrones tolerantes. En 1975 se estableció el Programa de Mejora Varietal de Cítricos
Los responsables de la dispersión de CTV antes de 1975 en España fueron varios factores: ausencia de material libre de virus, tráfico de material vegetal infectado (alta infección en algunas zonas), sobreinjerto, introducciones clandestinas y los vectores, aunque el principal responsable de la distribución de tristeza fue el hombre
A patir de 1975 con el Programa de Mejora Varietal de Cítricos, la puesta a punto de la técnica ELISA para CTV en 1979, el establecimiento de parcelas para estudios epidemiológicos y realización de prospecciones a gran escala, cambiaron la situación siendo a partir de 1990 los responsables de la dispersión de CTV, los vectores, Aphis gossypii, Aphis spiraecola y Toxoptera auranti
Un aspecto fundamental fue que a partir de 1989 se realizó una continua divulgación de recomendaciones para la reconversión citrícola española, y en 2003 el 80% de la citricultura que son unos 105 millones de árboles provenía de árboles libres de virus injertados sobre patrones tolerantes a la tristeza
La sharka tipo Marcus una gran amenaza para el cultivo de melocotonero (Autora: A.B. Ruiz-García)
Este virus (Plum pox virus - PPV) es el único virus de Prunus transmitido de forma natural por pulgones. La enfermedad es de cuarentena por lo que la producción y movimiento de material vegetal ha de ser controlado legalmente. La sintomatología de la enfermedad se traduce en una pérdida de calidad del fruto, así como una caída prematura de los mismos.


El virus pertenece a la familia Potyviridae y concretamente al género Potyvirus con una organización genómica típica de este género. El genoma está compuesto por una molécula de ARN de cadena simple con 9.741- 9.795 nucleótidos que tiene una proteína unida a su extremo 5’ (VPg) y una cola poliA en el extremo 3’. Los viriones son partículas flexuosas y filamentosas (750 x 15 nm).
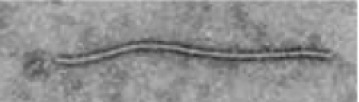
El ARN genómico se traduce en una poliproteína que se procesa proteolíticamente por tres proteasas codificadas por el propio virus, que dan lugar una única proteína de cubierta y varias proteínas no estructurales asociadas a la replicación, al movimiento del virus y a su transmisión por pulgones.
La facilidad de transmitirse por pulgón y por multiplicación vegetativa obliga a medidas técnicas y legales de control de viveros. La enfermedad no mata los árboles infectados por lo que hay un reservorio permanente si no se arrancan los árboles infectados, y en variedades susceptibles las pérdidas pueden alcanzar el 100% de la producción.
Un estudio realizado para estimar los costes asociados a la enfermedad reveló que el control de la sharka ha requerido más de 10.000 millones € sólo entre el periodo comprendido entre 1976 y 2006 (Cambra et al., 2006. Bull. EPPO 36, 202-204).
Entre las razas del virus el tipo Marcus (PPV-M) es el más agresivo y problemático, puesto que tiene una fácil transmisión por pulgones desde cualquier Prunus a otros Prunus. Además se dispersa rápidamente entre melocotoneros y su sintomatología es más agresiva que cualquiera de las otras razas descritas hasta el momento. La presencia de este tipo de virus en zonas de cultivo de melocotonero puede tener un enorme impacto, tan alto, que la producción puede verse mermada drásticamente.
El control de PPV requiere la producción de plantas de vivero en zonas libres de PPV, evitar la introducción de aislados PPV-M agresivos en melocotonero, realizar prospecciones para la detección precoz de arboles infectados por PPV-M y erradicación. Por último es necesario se debe profundizar en los programas de mejora de la resistencia y realizar cultivo bajo malla antipulgón.
Para evitar el problema de la sharka tipo Marcus se requiere de forma imprescindible la colaboración entre el sector, los productores y viveristas y la Administración, Servicios de Sanidad Vegetal, Laboratorios de Referencia y políticos.
Un poco de historia: epidemiología del virus de la sharka en España (Autora: A.B. Ruiz-García)

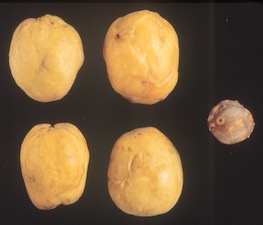
El cultivar protegido ‘Red Beaut’ PPV-infectados se distribuyeron legal e ilegalmente en todas las áreas de producción temprana de frutales de hueso-carozo, debido a su alto interés comercial. (1980-1986).
A pesar de los esfuerzos del Ministerio de Agricultura, fue imposible convencer a los agricultores de la necesidad de destruir los árboles infectados y erradicar una enfermedad que estaba causando ligeras pérdidas en frutos con alto valor comercial en esa época (0.8 – 1 €/kg).
…además, en aquel tiempo la técnica ELISA con anticuerpos policlonales de baja especificidad, no era fiable para detección y erradicación

‘Red Beaut’ se convirtió en una importante fuente de inóculo de PPV y los pulgones vectores dispersaron muy eficazmente el virus a otros ciruelos japoneses y albaricoqueros (damascos)

Sensibilidad a PPV-D de 24 diferentes cultivares de ciruelo japonés (Los síntomas en hojas se clasificaron en tres grupos):
Síntomas obvios:
606
Red Beaut
Ambra
Black Beaut
Durado
Santa Rosa
Sierra Plum
Golden Japan
Fortune
Autum Giant
Síntomas ligeros
Black Amber
Delbarazur (Strival)
Friar
Freedon (Larry Ann)
Ozark Premier
Wickson
Formosa
Simka
Síntomas muy suaves o sin síntomas
Carolina Harris
Catalina
Beauty (Nueva Extremadura)
Queen Rosa
Midnight Sun
Laroda
Sun Gold
Superior Angeleno
Superior Black Gold
Trompellot 1 and 2
Susy
Superior Black Diamond
Royal Diamond
Pioneer
- Los síntomas de PPV-D en hojas no se correlacionan con aquellos en frutos.
- La mayoría de los cultivares de P. salicina no muestran síntomas en frutos o los muestran muy ligeros afectando sólo a la piel.
- Los frutos desechados de árboles infectados únicamente suponen como máximo el 15%. Ello hace posible un beneficio económico incluso en plantaciones con alta incidencia de la enfermedad de la sharka.
...la producción de damascos (albaricoques) tempranos fue severamente dañada en todas las zonas mediterráneas de producción.
Clasificación de los virus vegetales de RNA de polaridad positiva (Autora: A.B. Ruiz-García)
Los más representativos se encuentran en las siguientes familias y géneros:
Familia Secoviridae:
- Género Cheravirus
- Género Swadavirus
- Género Sequivirus
- Género Torradovirus
- Género Waikivirus
- Subfamilia Comovirinae
- Género Comovirus
- Género Fabavirus
- Género Nepovirus
Familia Bromoviridae:
- Género Alfamovirus
- Género Anulavirus
- Género Bromovirus
- Género Ilarvirus
- Género Oleavirus
Familia Benyaviridae:
- Género Benyvirus
Familia Botourmiarividae:
- Género Botlivirus
- Género Magolivirus
- Género Ourmiavirus
- Género Scleroulivirus
Familia Closteroviridae
- Género Ampelovirus
- Género Closterovirus
- Género Crinivirus
- Género Velarivirus
Familia Kitaviridae
- Género Blunervirus
- Género Cilevirus
- Género Higrevirus
Familia Luteoviridae
- Género Enamovirus
- Género Luteovirus
- Género Polerovirus
Familia Potyviridae
- Género Bremovirus
- Género Brambyvirus
- Género Bymovirus
- Género Ipomovirus
- Género Macluravirus
- Género Poacevirus
- Género Potyvirus
- Género Roymovirus
- Género Rymovirus
- Género Tritimovirus
Familia Solemoviridae
- Género Polemovirus
- Género Sobemovirus
Familia Tombusviridae
- Género Avenavirus
- Subfamilia Calvusvirinae
- Género Umbravirus
- Subfamilia Procedovirinae
- Género Alphacarmovirus
- Género Alphanecrovirus
- Género Aureusvirus
- Género Betacarmovirus
- Género Betanecrovirus
- Género Gallantivirus
- Género Gammacarmovirus
- Género Macanavirus
- Género Machlomovirus
- Género Panicovirus
- Género Pelarspovirus
- Género Tombusvirus
- Género Zeavirus
- Subfamilia Regressovirinae
- Género Dianthovirus
Familia Virgaviridae
- Género Furovirus
- Género Goravirus
- Género Hordeivirus
- Género Pecluvirus
- Género Pomovirus
- Género Tobamovirus
- Género Tobravirus
Géneros no asignados a familias
- Género Albetovirus
- Género Aumaivirus
- Género Idaeovirus
- Género Papanivirus
- Género Sinaivirus
- Género Virtovirus
Familia Alphaflexiviridae
- Género Allexivirus
- Género Botrexvirus
- Género Lolavirus
- Género Mandarivirus
- Género Platypuvirus
- Género Potexvirus
- Género Sclerodarnavirus
Familia Betaflexiviridae
- Subfamilia Quinvirinae
- Género Carlavirus
- Género Foveavirus
- Género Robigovirus
- Subfamilia Trivirinae
- Género Capillovirus
- Género Chordovirus
- Género Citrivirus
- Género Divavirus
- Género Prunevirus
- Género Tepovirus
- Género Trichovirus
- Género Vitivirus
- Género Wamavirus
Familia Gammaflexiviridae
- Género Mycoflexivirus
Familia Deltaflexiviridae
- Género Deltaflexivirus
Familia Tymoviridae
- Género Maculavirus
- Género Marafivirus
- Género Tymovirus
Toma de muestras y expedición de muestras (Autora: A.B. Ruiz-García)
TOMA DE MUESTRAS
Toma de muestras muy dependiente de la especie y período vegetativo
Reposo invernal o latencia: dardos, yemas corteza, ramas, madera de poda o planta entera.
Período vegetativo:
Primavera: flores, hojas expandidas, dardos brotados, ramilletes de mayo, planta entera.
Verano: hojas, frutos, planta entera.
Otoño: hojas, yemas.
EXPEDICIÓN DE MUESTRAS
Tomar muestras y conservar a 4ºC (máximo 3 días).
Empaquetar en papel humedecido e incluir en bolsa de plástico agujereada (que permita ligera aireación).
Señalar en el paquete: “Contiene muestras vegetales vivas”.
Rellenar e incluir hoja del Laboratorio de Referencia.
Remitir a principios de semana por correo urgente, con dirección e identificación clara, incluída la del remitente.
Características de las virosis vegetales (Autora: A.B. Ruiz-García)
- SON FRECUENTES (Fácil infección, tiempo exposición largo especialmente en plantas leñosas ...)
- HAY GRANDES POBLACIONES HOMOGÉNEAS DE PLANTAS CULTIVADAS (Inmóviles)
- SON INCURABLES (No sistema inmune) Y ES IMPOSIBLE UN CONTROL QUÍMICO
- SON TRANSMISIBLES: vegetativamente (injerto, multiplic. vegetativa), semilla, polen, vectores...
Esto hace que la PREVENCIÓN / ERRADICACIÓN / SELECCIÓN SANITARIA sean las únicas posibilidades eficaces hoy en día para su control.
Es importante conocer la taxonomía y clasificación de virus y viroides para poder mitigar sus efectos. El comité internacional de taxonomía de virus (ICTV) es el encargado de su clasificación. Así y siguiendo sus indicaciones los virus se pueden clasificar en base las características de sus ácidos nucleicos.
Hay más de 700 virus de plantas conocidos y cada día aumenta el número.
Alrededor del 69% se transmiten por vectores:
- 55% transmitidos por hemípteros siendo el 72% de estos, pulgones. Aproximadamente 275 especies de virus pertenecientes a 19 géneros y un 11% mosca blanca
- 13% transmitidos por otros insectos (Coleoptera, Thysanoptera, Ortoptera, Dermaptera, Lepidoptera, Diptera, Heteroptera)
- 2% son vectores no insectos (ácaros, nematodos, hongos, cuscuta)
- 30% contacto, semilla o polen
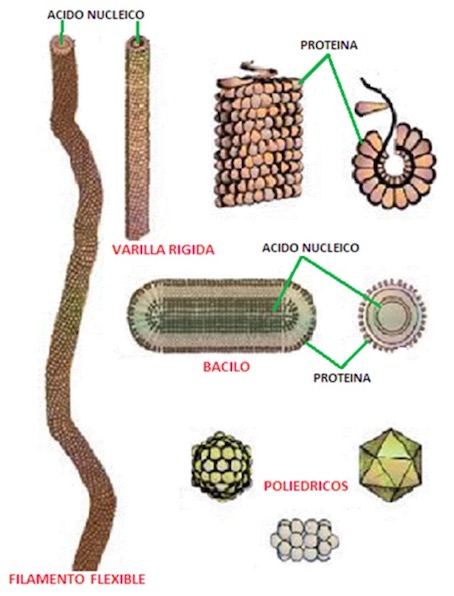
¿Qué es un virus? (Autora: A.B. Ruiz-García)
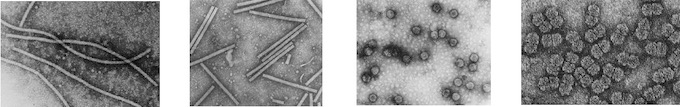
Los virus son organismos muy pequeños que no pueden ser vistos con microscopio óptico
Se multiplican únicamente en células vivas y muchos de ellos producen enfermedad
Están formados por:
ácido nucleico – contiene información genético
proteína – forma la cubierta protectora o cápsida
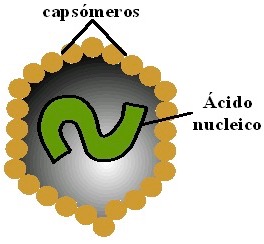
El ácido nucleico de la mayoría de los virus fitopatógenos es de RNA, aunque algunos son de DNA.
Tipos de virus
Virus helicoidales:
En forma de varilla rígida o de filamento flexible. Los primeros miden alrededor de 15 nanómetros de ancho por 300 de largo; los filamentos flexibles por lo sobre 10 a 13 nanómetros de ancho y de 480 a 2000 nanómetros de longitud
En forma de bacilo o barras cilíndricas, son de tres a cinco veces más largos que su anchos, midiendo entre 52-75 por 300-380 nanómetros
Virus poliédricos:
La mayoría de estos virus son en realidad icosaédricos, varían en diámetro entre 17 a 60 nanómetros
Los virus penetran en las plantas únicamente a través de
Heridas hechas mecánicamente
Vectores
Grano de polen infectado que se deposita en un óvulo
Para que la infección se lleve a cabo el virus debe moverse de una célula a otra y debe multiplicarse en ellas. El movimiento de célula a célula se lleva a cabo a través de los plasmodesmos que comunican células adyacentes
Cuando el virus llega al floema es transportado a gran distancia y se disemina sistémicamente por toda la planta
La distribución del virus dentro de la planta varía según el virus y la especie de planta
La transmisión de los virus vegetales puede ser:
- Propagación vegetativa
- Mecánicamente
- Semilla
- Polen
- Vectores: Insectos, Ácaros, Nematodos, Hongos, Cuscuta
 orcid.org/0000-0001-8406-7963
orcid.org/0000-0001-8406-7963