¿Qué es la secuencación masiva? (Autora: A.B. Ruiz-García)
05/08/18 17:42 ..... Divulgacion
La secuenciación masiva (NGS o HTS) es una tecnología que permiten la secuenciación simultánea de millones de secuencias de ácidos nucleicos presentes en una muestra.
Esto es posible gracias al avance de la tecnología que ha permitido el desarrollo de equipos capaces de realizar de forma simultánea la secuenciación de DNA de millones de fragmentos diferentes de ADN.
Así pues, empleando esta tecnología, es potencialmente posible la detección de cualquier virus que esté presente en una muestra de una planta.

Esta tecnología puede usarse para reemplazar una amplia gama de métodos convencionales, ya que además tiene potencial para identificación de virus tanto conocidos como desconocidos.
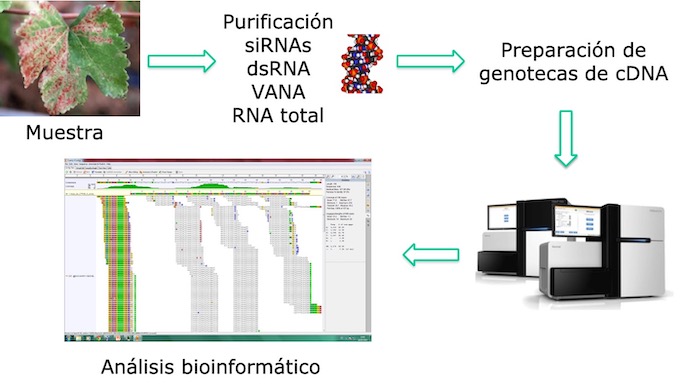
Básicamente el proceso es sencillo: 1) se selecciona una muestra; 2) se purifican los ácidos nucleicos de la muestra; 3) se preparan genotecas, es decir los ácidos nucleicos se trocean y se les añaden adaptadores para que sea posible realizar una reacción de PCR a la vez en todas los trozos con los adaptadores; 4) el equipo secuencia millones de estos fragmentos y 5) se realiza un análisis bioinformático para averiguar que virus hay presentes
En el campo de la virología vegetal, HTS está revolucionando la comprensión de los científicos sobre la naturaleza ubicua de los virus y demuestra claramente que apenas estamos comenzando a conocer la gran diversidad viral previamente desconocida.
Se puede aplicar para abordar diferentes campos de la virología:
-Etiología viral
-Caracterización de virus
-Taxonomía
-Genética de poblaciones
-Desarrollo de métodos de diagnóstico
En los últimos años se han descrito una gran cantidad de virus nuevos mediante el empleo de esta tecnología, se ha propuesto la creación de nuevos géneros y el conocimiento adquirido ha conllevado una continua revisión de la taxonomía.
Esto es posible gracias al avance de la tecnología que ha permitido el desarrollo de equipos capaces de realizar de forma simultánea la secuenciación de DNA de millones de fragmentos diferentes de ADN.
Así pues, empleando esta tecnología, es potencialmente posible la detección de cualquier virus que esté presente en una muestra de una planta.

Esta tecnología puede usarse para reemplazar una amplia gama de métodos convencionales, ya que además tiene potencial para identificación de virus tanto conocidos como desconocidos.
Básicamente el proceso es sencillo: 1) se selecciona una muestra; 2) se purifican los ácidos nucleicos de la muestra; 3) se preparan genotecas, es decir los ácidos nucleicos se trocean y se les añaden adaptadores para que sea posible realizar una reacción de PCR a la vez en todas los trozos con los adaptadores; 4) el equipo secuencia millones de estos fragmentos y 5) se realiza un análisis bioinformático para averiguar que virus hay presentes

En el campo de la virología vegetal, HTS está revolucionando la comprensión de los científicos sobre la naturaleza ubicua de los virus y demuestra claramente que apenas estamos comenzando a conocer la gran diversidad viral previamente desconocida.
Se puede aplicar para abordar diferentes campos de la virología:
-Etiología viral
-Caracterización de virus
-Taxonomía
-Genética de poblaciones
-Desarrollo de métodos de diagnóstico
En los últimos años se han descrito una gran cantidad de virus nuevos mediante el empleo de esta tecnología, se ha propuesto la creación de nuevos géneros y el conocimiento adquirido ha conllevado una continua revisión de la taxonomía.
blog comments powered by Disqus
 orcid.org/0000-0001-8406-7963
orcid.org/0000-0001-8406-7963