Adiciones sobre el micovirus Penicillimonavirus gammaplasmopara o Penicillimonavirus gammaplasmoparae (Autora: Ana B. Ruiz-García)
18/09/23 18:07 ..... Divulgacion
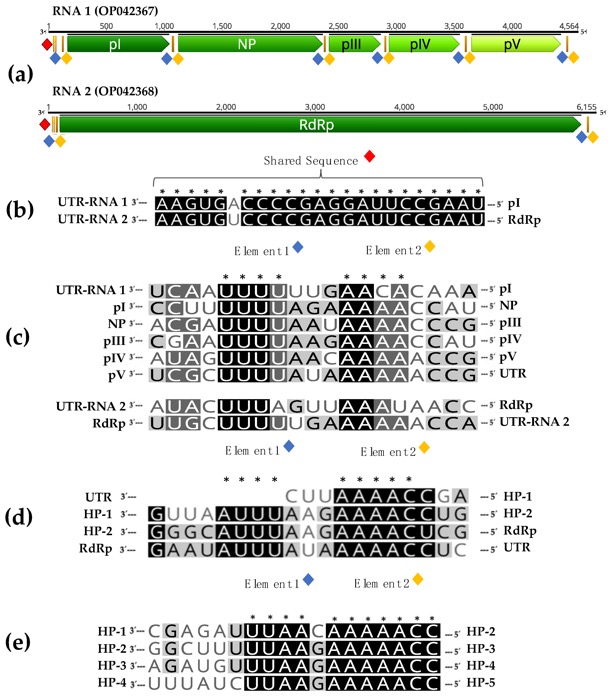
Como recordaréis en mayo del año pasado (este es el link de la comunicación) el Dr Félix Morán defendió su tesis doctoral y presentó el genoma bipartito de un micovirus, el Plasmopara-viticola-lesion-associated mononegaambi virus 3 que recientemente ha sido clasificado como Penicillimonavirus gammaplasmoparae, lo que supone como anticipábamos la primera especie del nuevo género Penicillimonarivus para el que hemos descrito la estructura genómica completa del virus.
Si queréis tener las secuencias de los dos RNA del virus podéis bajárosla de GenBank con los números de Acceso OP042367 y OP042368.
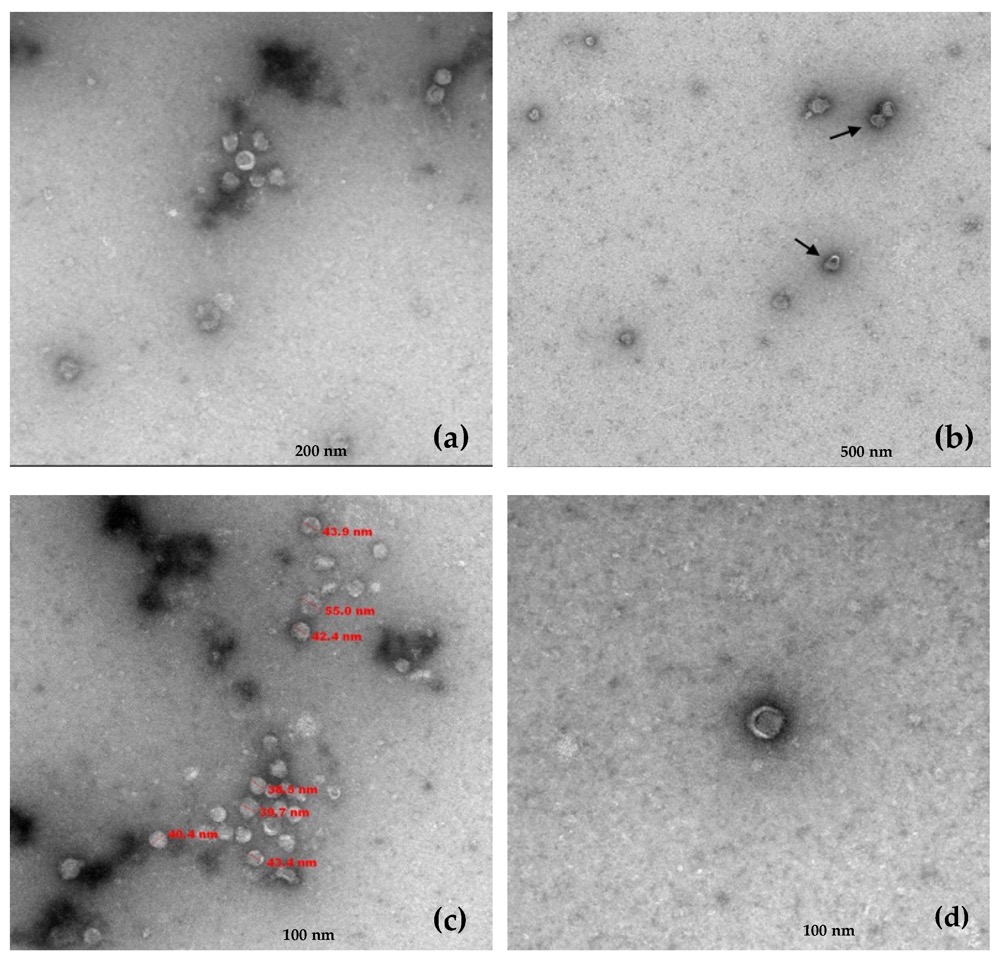
Para más información sobre el micovirus ahora podéis consultar nuestra publicación sobre los avances que hemos realizado en el artículo científico publicado en Plants. En el artículo podréis comprobar cómo es el virus cuando se observa por microscopio electrónica.
Si queréis tener las secuencias de los dos RNA del virus podéis bajárosla de GenBank con los números de Acceso OP042367 y OP042368.
Para más información sobre el micovirus ahora podéis consultar nuestra publicación sobre los avances que hemos realizado en el artículo científico publicado en Plants. En el artículo podréis comprobar cómo es el virus cuando se observa por microscopio electrónica.

Comments
Desarrollamos y validamos un nuevo duplex para la identificación del viroide latente del melocotonero en colaboración con el EURL de Virus (Autora: A.B. Ruiz-García)
27/04/23 17:29 ..... Divulgacion
El viroide del mosaico latente del melocotón (PLMVd) es un patógeno importante que causa enfermedades en los melocotones.
El control de este viroide sigue siendo problemático porque la mayoría de las variantes de PLMVd son asintomáticas y, aunque hay disponibles diversas técnicas de detección, la fiabilidad de los métodos basados en PCR se ve comprometida por la estructura secundaria compleja y ramificada de su ARN y por su diversidad genética.
En este estudio, se ha desarrollado y validado un método de RT-qPCR que potencialmente puede detectar todas las variantes conocidas de PLMVd. Además, para simplificar la preparación de la muestra, se ha puesto a punto un procedimiento rápido de extracción basados en el uso de extracto crudp o impresión de tejido. El rendimiento de este nuevo procedimiento se ha evaluado en un estudio interlaboratorio en el que han participado cinco laboratorios especializados.
Se ha demostrado que el nuevo método, en combinación con enfoques de preparación rápida de muestras, es factible y fiable, con la ventaja de detectar todos los diferentes aislados/variantes de PLMVd analizados en una sola reacción, lo que reduce los costos del diagnóstico de rutina.
Para detalles podeis consultar nuestro artículo publicado en la revista Plants “Development and Validation of a Duplex RT-qPCR for Detection of Peach Latent Mosaic Viroid and Comparison of Different Nucleic-Acid-Extraction Protocols”
El control de este viroide sigue siendo problemático porque la mayoría de las variantes de PLMVd son asintomáticas y, aunque hay disponibles diversas técnicas de detección, la fiabilidad de los métodos basados en PCR se ve comprometida por la estructura secundaria compleja y ramificada de su ARN y por su diversidad genética.
En este estudio, se ha desarrollado y validado un método de RT-qPCR que potencialmente puede detectar todas las variantes conocidas de PLMVd. Además, para simplificar la preparación de la muestra, se ha puesto a punto un procedimiento rápido de extracción basados en el uso de extracto crudp o impresión de tejido. El rendimiento de este nuevo procedimiento se ha evaluado en un estudio interlaboratorio en el que han participado cinco laboratorios especializados.
Se ha demostrado que el nuevo método, en combinación con enfoques de preparación rápida de muestras, es factible y fiable, con la ventaja de detectar todos los diferentes aislados/variantes de PLMVd analizados en una sola reacción, lo que reduce los costos del diagnóstico de rutina.
Para detalles podeis consultar nuestro artículo publicado en la revista Plants “Development and Validation of a Duplex RT-qPCR for Detection of Peach Latent Mosaic Viroid and Comparison of Different Nucleic-Acid-Extraction Protocols”
Revisión del nuevo estándar de la EPPO para el diagnóstico de CTV (Autora: A. B. Ruiz-García)
29/03/23 21:22 ..... Divulgacion
El estándar del diagnóstico para CTV ha sido revisado y publicado por la EPPO. Nuestro equipo ha participado en la redacción y modificación del estándar que incluye además el diagnóstico diferencial de razas agresivas de CTV.
Para más información podéis consultar la publicación PM7 31(2) Citrus tristeza virus
Para más información podéis consultar la publicación PM7 31(2) Citrus tristeza virus
Desarrollamos el método más inclusivo para la detección del virus del enrrollado 2 (Autora: A.B. Ruiz-García)
16/02/23 16:35 ..... Divulgacion
El virus del enrollado de la vid 1 (GLRaV-1) está asociado a la enfermedad del enrollado de la hoja de la vid y está considerado a nivel nacional y europeo como un patógeno que debe estar ausente en el material vegetal de propagación. Por este motivo, es de gran importancia disponer de técnicas de detección específicas, sensibles y fiable. Esta especie se caracteriza por su enorme variabilidad y por tanto en la dificultad de disponer de un método que detecte las variantes del virus. En nuestro laboratorio hemos desarrollado un método de detección de GLRaV-1 basado en una RT-PCR cuantitativa en tiempo real TaqMan que además incluye un control interno del huésped en una reacción dúplex. El método ha sido validado siguiendo los estándares de la EPPO y aplicado para el diagnóstico de material vegetal de campo y vectores de transmisión. Este nuevo protocolo ha resultado ser muy sensible y mucho más específico que los métodos actualmente disponibles para la detección y cuantificación absoluta del título viral GLRaV-1 con la capacidad de detectar todos los aislados descritos hasta el momento.
Para más detalles os invitamos a leer el artículo publicado en la revista Plants con la descripción del trabajo
“A Novel and Highly Inclusive Quantitative Real-Time RT-PCR Method for the Broad and Efficient Detection of Grapevine Leafroll-Associated Virus 1”
Para más detalles os invitamos a leer el artículo publicado en la revista Plants con la descripción del trabajo
“A Novel and Highly Inclusive Quantitative Real-Time RT-PCR Method for the Broad and Efficient Detection of Grapevine Leafroll-Associated Virus 1”
Descubrimos el viroide del hoyuelo de la manzana (ADFVd) en granado (Autora: A.B. Ruiz-García)
12/01/23 13:22 ..... Divulgacion
En un trabajo reciente hemos descubierto que el viroide del hoyuelo de la manzana está presente en granado.
Este estudio se inició cuando se recibieron en el laboratorio muestras con una sintomatología muy particular, tal como se muestra en la figura de abajo.
Figura 1. Granada con síntomas de anillos cloróticos. Muestra infectadas por el viroide del hoyuelo de la manzana - apple dimple fruit viroid (ADFVd).

Para la identificación del agente etiológico del posible agente causal de esta sintomatología se realizó secuenciación masiva a partir del ARN total de la muestra.
Los resultados mostraron la presencia del viroide del hoyuelo del manzano.
Al realizar prospecciones se observaron frutos con esta sintomatología como se muestra en la figura 2
Figura 2

La presencia del viroide se puede confirmar mediante RT-PCR con los iniciadores que diseñados en el estudio.
ADFVd F3: 5′-TTGACTAGATGCCCGCCTGAC-3′ y ADFVd R3 5′-CACGACCTAGAGCCGCCTCCA-3′ or ADFVd F4: 5′-AAGGACTTGACTAG-ATGCCCGCCTG-3 y ADFVd R4: 5′ AGTCATTAGCGCAGGGGGGTGC-3′.
Este estudio se inició cuando se recibieron en el laboratorio muestras con una sintomatología muy particular, tal como se muestra en la figura de abajo.
Figura 1. Granada con síntomas de anillos cloróticos. Muestra infectadas por el viroide del hoyuelo de la manzana - apple dimple fruit viroid (ADFVd).

Para la identificación del agente etiológico del posible agente causal de esta sintomatología se realizó secuenciación masiva a partir del ARN total de la muestra.
Los resultados mostraron la presencia del viroide del hoyuelo del manzano.
Al realizar prospecciones se observaron frutos con esta sintomatología como se muestra en la figura 2
Figura 2

La presencia del viroide se puede confirmar mediante RT-PCR con los iniciadores que diseñados en el estudio.
ADFVd F3: 5′-TTGACTAGATGCCCGCCTGAC-3′ y ADFVd R3 5′-CACGACCTAGAGCCGCCTCCA-3′ or ADFVd F4: 5′-AAGGACTTGACTAG-ATGCCCGCCTG-3 y ADFVd R4: 5′ AGTCATTAGCGCAGGGGGGTGC-3′.
Estudios de HTS revelan la diversidad del virus del mosaico del pepino (CMV) en Eslovaquia y nuevos huéspedes de CMV de la familia Papaveraceae (Autora: A.B. Ruiz-García)
25/06/22 17:35 ..... Divulgacion
El virus del mosaico del pepino (CMV; Cucumovirus, Bromoviridae) es un virus omnipresente caracterizado por una amplia gama de huéspedes y una alta variabilidad genética.
Mediante secuenciación masiva, hemos caracterizado genomas completos de 14 variantes de CMV de diferentes plantas hospedadoras. De estas, tres variantes procedían de especies de Papaveraceae (amapola oleaginosa, amapola común y celidonia mayor), previamente poco descritas como huéspedes naturales del CMV. Los aislados de CMV estudiados y que eran de Eslovaquia se pueden dividir en dos grupos genéticamente diferentes, Ia y II, respectivamente.
La variante SL50V, caracterizada por una secuencia de ARN2 divergente, representa potencialmente una variante recombinante.
En cuatro muestras (T101, SL50V, CP2, MVU2-21), se identificó la presencia de ARN de CMV satélite junto con CMV. Aunque se transmitió mecánicamente a plantas de pepino experimentales, no se pudo establecer el papel del ARN satélite en la sintomatología observada debido a una infección compleja de los huéspedes originales con diferentes virus.
Para más detalles podéis consultar el artículo “High-Throughput Sequencing Discloses the Cucumber Mosaic Virus (CMV) Diversity in Slovakia and Reveals New Hosts of CMV from the Papaveraceae Family”

Mediante secuenciación masiva, hemos caracterizado genomas completos de 14 variantes de CMV de diferentes plantas hospedadoras. De estas, tres variantes procedían de especies de Papaveraceae (amapola oleaginosa, amapola común y celidonia mayor), previamente poco descritas como huéspedes naturales del CMV. Los aislados de CMV estudiados y que eran de Eslovaquia se pueden dividir en dos grupos genéticamente diferentes, Ia y II, respectivamente.
La variante SL50V, caracterizada por una secuencia de ARN2 divergente, representa potencialmente una variante recombinante.
En cuatro muestras (T101, SL50V, CP2, MVU2-21), se identificó la presencia de ARN de CMV satélite junto con CMV. Aunque se transmitió mecánicamente a plantas de pepino experimentales, no se pudo establecer el papel del ARN satélite en la sintomatología observada debido a una infección compleja de los huéspedes originales con diferentes virus.
Para más detalles podéis consultar el artículo “High-Throughput Sequencing Discloses the Cucumber Mosaic Virus (CMV) Diversity in Slovakia and Reveals New Hosts of CMV from the Papaveraceae Family”

Caracterizamos por primera vez un micovirus asociado al oidio de la vid y averiguamos que consta de un genoma bipartito (Autores: A.B. Ruiz-García y A. Olmos)
19/05/22 21:00 ..... Divulgacion
La tesis doctoral de Félix Morán Villamizar que hemos dirigido en el LNR de virus de leñosas por la Dra. Ana B. Ruiz-García y por el Dr. Antonio Olmos, ha sido presentada y defendida hoy en la Universidad de Valencia. Enhorabuena al Dr. Félix Morán Villamizar.
Entre otras cosas hemos presentado por primera la secuencia completa del virus Plasmopara-viticola-lesion-associated mononegaambi virus 3, que representa la primera especie de un nuevo género para la que hemos determinado su estructura genómica que ha resultado ser bipartita.
De hecho en el laboratorio hemos identificamos el virus Plasmopara-viticola-lesion-associated mononegaambi virus 3, un mymonavirus asociado a hongos, en plantas de vid que muestran una sintomatología inusual de curvatura hacia arriba en las hojas y un deterioro prematuro.

La familia Mymonaviridae es una familia que comprende virus de ARN monocatenario de sentido negativo que infectan hongos filamentosos, que se han asociado a oomicetos, plantas e insectos.
Aunque la primera descripción del genoma de mymonavirus se realizó hace una década, la organización y estructura del genoma los géneros de la familia, no ha está clara hasta la fecha.
En el laboratorio hemos determinado el genoma completo del mymonavirus Plasmopara-viticola-lesion-associated mononegaambi virus 3 que representa la primera secuencia genómica completa de un posible nuevo género.
Además, proporcionamos pruebas sólidas de que el genoma de es bipartito y comprende dos moléculas de ARN de alrededor de 6150 y 4560 nt.
Nuestros resultados indican que el patógeno oídio, Erysiphe necator, también está presente en las plantas analizadas y sugieren que este virus puede estar infectando este hongo.
Sin embargo, aún está por determinar si el hongo y/o el micovirus están asociados con la sintomatología que inicialmente motivó estos estudios.
Entre otras cosas hemos presentado por primera la secuencia completa del virus Plasmopara-viticola-lesion-associated mononegaambi virus 3, que representa la primera especie de un nuevo género para la que hemos determinado su estructura genómica que ha resultado ser bipartita.
De hecho en el laboratorio hemos identificamos el virus Plasmopara-viticola-lesion-associated mononegaambi virus 3, un mymonavirus asociado a hongos, en plantas de vid que muestran una sintomatología inusual de curvatura hacia arriba en las hojas y un deterioro prematuro.

La familia Mymonaviridae es una familia que comprende virus de ARN monocatenario de sentido negativo que infectan hongos filamentosos, que se han asociado a oomicetos, plantas e insectos.
Aunque la primera descripción del genoma de mymonavirus se realizó hace una década, la organización y estructura del genoma los géneros de la familia, no ha está clara hasta la fecha.
En el laboratorio hemos determinado el genoma completo del mymonavirus Plasmopara-viticola-lesion-associated mononegaambi virus 3 que representa la primera secuencia genómica completa de un posible nuevo género.
Además, proporcionamos pruebas sólidas de que el genoma de es bipartito y comprende dos moléculas de ARN de alrededor de 6150 y 4560 nt.

Nuestros resultados indican que el patógeno oídio, Erysiphe necator, también está presente en las plantas analizadas y sugieren que este virus puede estar infectando este hongo.
Sin embargo, aún está por determinar si el hongo y/o el micovirus están asociados con la sintomatología que inicialmente motivó estos estudios.
Estudios en el viroma del olivo en España, el olivavirus OLYaV (Autora: A.B. Ruiz-García)
12/01/22 13:22 ..... Divulgacion
En un reciente trabajo el uso de secuenciación masiva (HTS) para el análisis de olivos españoles que mostraban decoloración de hojas y amarilleamiento, la defoliación y/o el declive ha proporcionado nuevos conocimientos sobre los virus de olivo presentes en España y ha abierto debates sobre las ventajas e inconvenientes de estas tecnologías con fines diagnósticos (Ruiz-García AB, Canales C, Morán F, Ruiz-Torres M, Herrera-Mármol M, Olmos A. Characterization of Spanish Olive Virome by High Throughput Sequencing Opens New Insights and Uncertainties. Viruses. 2021 Nov 6;13:2233). En este estudio describimos por primera vez en el olivar español la presencia del virus asociado al amarillamiento de la hoja del olivo (OLYaV), un virus cuya secuencia completa fue obtenida por nuestro grupo previamente y que ofrece información para la creación de un nuevo género (Olivavirus) en la familia Closteroviridae (véase la comunicación del 17/10/2020).
En la figura 1 se muestra la sintomatología observada en campo en árboles de olivo.

Figura 1. Síntomas de defoliación, muerte y amarilleamiento de hojas causados por OLYaV.
La comparación entre las secuencias nucleotídica/proteínica del nuevo aislado español de OLYaV y las secuencias nucleotídica/proteínica del aislado brasileño previamente secuenciado por nuestro equipo también, se muestra en la figura 2.
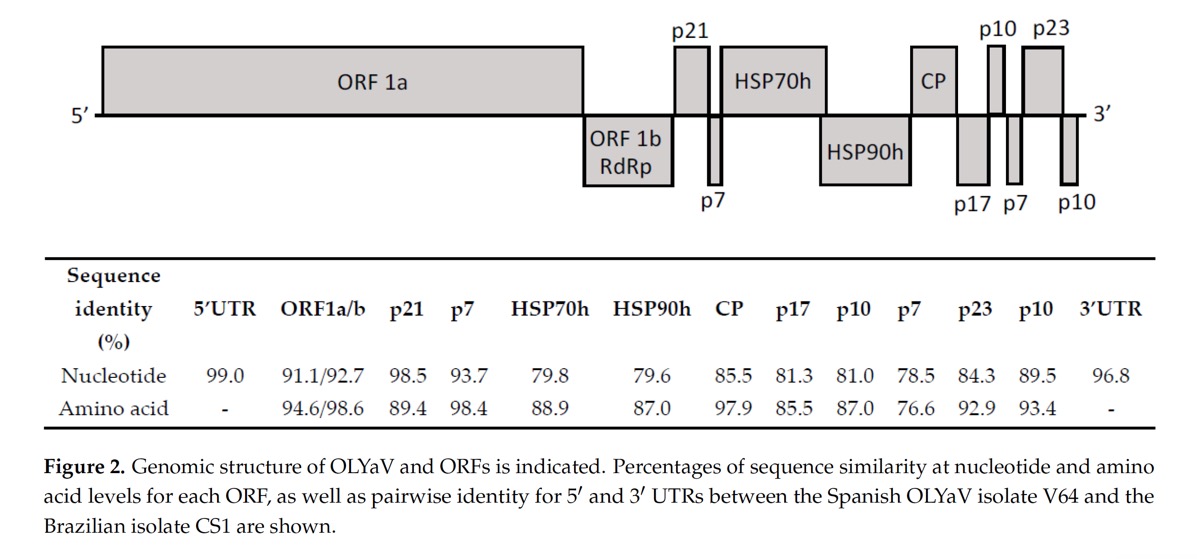
Como se puede observar las regiones más óptimas para la detección del virus deberán basarse en la región 5’UTR, p21 y/o 3’UTR y no la HSP70 que es la que actualmente se emplea y tiene un bajo porcentaje de homología (79.8%) lo que implica la presencia de falsos negativos porque la técnica puede no cubrir la variabilidad del virus.
Además se ha obtenido la segunda secuencia completa de OLYaV. Este virus también se ha detectado en un posible vector, el psílido Euphyllura olivina. Además, se ha confirmado en España también la presencia de Olea europaea geminivirus (OEGV), recientemente informado en Italia, y se ha obtenido la secuencia completa de dos aislados por secuenciación HTS y por secuenciación Sanger.
En la figura 1 se muestra la sintomatología observada en campo en árboles de olivo.

Figura 1. Síntomas de defoliación, muerte y amarilleamiento de hojas causados por OLYaV.
La comparación entre las secuencias nucleotídica/proteínica del nuevo aislado español de OLYaV y las secuencias nucleotídica/proteínica del aislado brasileño previamente secuenciado por nuestro equipo también, se muestra en la figura 2.

Como se puede observar las regiones más óptimas para la detección del virus deberán basarse en la región 5’UTR, p21 y/o 3’UTR y no la HSP70 que es la que actualmente se emplea y tiene un bajo porcentaje de homología (79.8%) lo que implica la presencia de falsos negativos porque la técnica puede no cubrir la variabilidad del virus.
Además se ha obtenido la segunda secuencia completa de OLYaV. Este virus también se ha detectado en un posible vector, el psílido Euphyllura olivina. Además, se ha confirmado en España también la presencia de Olea europaea geminivirus (OEGV), recientemente informado en Italia, y se ha obtenido la secuencia completa de dos aislados por secuenciación HTS y por secuenciación Sanger.
El níspero como nuevo huesped del viroide del manzano de cabeza de martillo (Autora: A.B. Ruiz-García)
16/12/21 12:32 ..... Divulgacion
En prospecciones de níspero en zonas productoras se detectó por primera vez como huésped alternativo un viroide que había sido descrito infectando manzano. El viroide del manzano de cabeza de martillo (AHVd). El viroide se identificó en una muestra de níspero la SL73.6 que se analizó por secuenciacón masiva. Entre los contigs relacionados con virus/viroides ensamblados a partir de datos SL73.6 HTS, se encontraron dos contigs de 489 y 376 nt relacionados con el viroide AHVd. El análisis bioinformático de estos contigs permitió la recuperación de un genoma circular completo de 376 nt cubierto por 7014 lecturas (cobertura promedio 2515.5x), el aislado AHVd-SL73.6 (depositado en GenBank, número de acceso OK272503). Para confirmar la presencia de AHVd en esta muestra, el genoma completo se secuenció mediante Sanger tras la amplificada por RT-PCR utilizando cebadores adyacentes de opuesta polaridad. Se obtuvo un producto de 376 pb, que se clonó y al secuenciarlo mediante Sanger, se confirmó la secuencia obtenida por HTS al 100%. Estos resultados muestran por primera vez la presencia de AHVd infectando al níspero, identificando así a este cultivo como un nuevo huésped de esta especie de viroide. El aislado de níspero español AHVd mostró una alta identidad de nucleótidos (93,97%) con el aislado de referencia (NC_028132, KR605506) de China, aunque el genoma AHVd-SL73.6 fue más corto (376 nt frente a 434 nt) debido a una eliminación de 56 nt en la posición genómica 55-110 y una deleción de 2 nt en la posición genómica 419-420 con respecto a la secuencia de referencia. Sin embargo, la estructura secundaria predicha para AHVd-SL73.6 mostró un plegamiento similar a los descritos previamente, resultando en una conformación característica de Pelamoviroides compuestos por un dominio en forma de barilla que contiene los nucleótidos que forman la estructura de cabeza de martillo y un dominio multiramificado.
En la siguiente figura se observa la estructura secundaria del viroide aislado de níspero.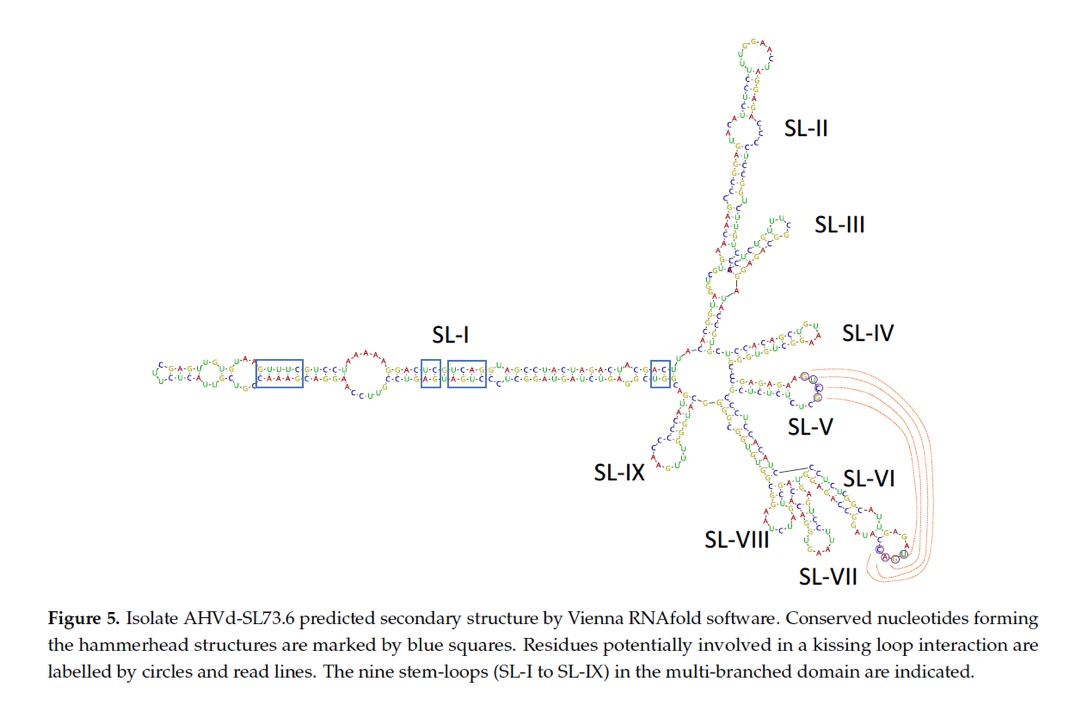
En la siguiente figura se observa la estructura secundaria del viroide aislado de níspero.

Primeras detecciones de la falsa sharka en níspero en España (Autora: A.B. Ruiz-García)
03/11/21 11:26 ..... Divulgacion
Continuando con el níspero y avanzar en el conocimiento de su estado sanitario en España, en prospecciones realizadas en áreas productoras de níspero se ha detectado por primera la presencia del virus de la falsa sharka, apple clorotic leaf spot virus (ACLSV) en níspero con una alta incidencia del 31.76%.
La secuencia y caracterización genómica del primer aislado español de ACLSV infectando níspero se realizó de la siguiente forma.
El análisis por secuenciación masiva (HTS) realizado en el ARN total extraído de la muestra SL73.6 rindió 31.867.726 lecturas d con un tamaño promedio de 134,8 nt. Se realizó la sustracción del genoma del níspero, tras el control de calidad de las lecturas y eliminar los adaptadores, lo que resultó en 1.698.300 lecturas que se utilizaron para el ensamblado de novo que dio lugar a 13.797 contigs. Se anotaron los contigs relacionados con virus
y viroides mediante BLASTN/X. Este análisis mostró 11 contigs de tamaños entre
6511 y 281 nt relacionados con ACLSV, confirmando la presencia de ACLSV en la muestra. La extensión de los contigs realizando el mapeo de las lecturas contra los contigs permitió la recuperación de un genoma casi completo de 7533 nt, cubierto por 44 039 lecturas (cobertura promedio 857,2x), aislado llamado SL73.6 y depositado en GenBank, número de acceso OK272502. Este aislado mostró el mayor porcentaje de identidad a nivel nueclotídico (83,7%) con el aislado alemán 38/85-B (KX579123) de manzano. El mapa de la organización del genoma SL73.6, la cobertura promedio de HTS y la similitud de proteínas de sus ORF con el aislado de ACLSV más cercano (KX579123) se muestran en la Figura1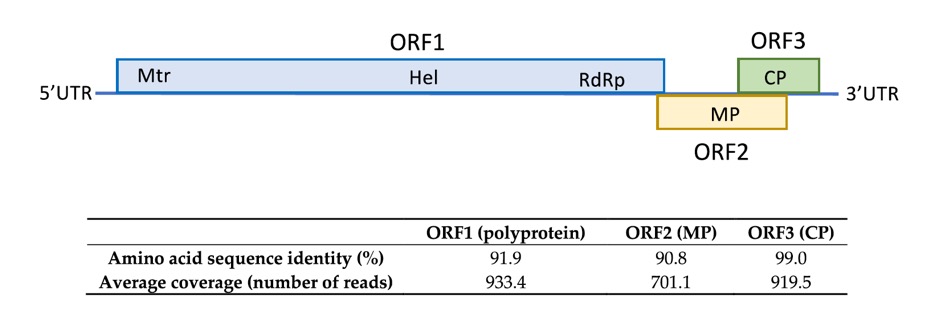
Figura 1. Similaridad aminoacídica del aislado SL73.6 con los ORFs del aislado de ACLSV más próximo (KX579123). Se muestra la cobertura por HTS.
En la siguiente figura se muestra la relación filogenética del aislado de ACLSV de níspero con otros aislados de ACLSV de otros orígenes y de otros hospedados.

La secuencia y caracterización genómica del primer aislado español de ACLSV infectando níspero se realizó de la siguiente forma.
El análisis por secuenciación masiva (HTS) realizado en el ARN total extraído de la muestra SL73.6 rindió 31.867.726 lecturas d con un tamaño promedio de 134,8 nt. Se realizó la sustracción del genoma del níspero, tras el control de calidad de las lecturas y eliminar los adaptadores, lo que resultó en 1.698.300 lecturas que se utilizaron para el ensamblado de novo que dio lugar a 13.797 contigs. Se anotaron los contigs relacionados con virus
y viroides mediante BLASTN/X. Este análisis mostró 11 contigs de tamaños entre
6511 y 281 nt relacionados con ACLSV, confirmando la presencia de ACLSV en la muestra. La extensión de los contigs realizando el mapeo de las lecturas contra los contigs permitió la recuperación de un genoma casi completo de 7533 nt, cubierto por 44 039 lecturas (cobertura promedio 857,2x), aislado llamado SL73.6 y depositado en GenBank, número de acceso OK272502. Este aislado mostró el mayor porcentaje de identidad a nivel nueclotídico (83,7%) con el aislado alemán 38/85-B (KX579123) de manzano. El mapa de la organización del genoma SL73.6, la cobertura promedio de HTS y la similitud de proteínas de sus ORF con el aislado de ACLSV más cercano (KX579123) se muestran en la Figura1

Figura 1. Similaridad aminoacídica del aislado SL73.6 con los ORFs del aislado de ACLSV más próximo (KX579123). Se muestra la cobertura por HTS.
En la siguiente figura se muestra la relación filogenética del aislado de ACLSV de níspero con otros aislados de ACLSV de otros orígenes y de otros hospedados.

Primeras detecciones de apple stem grooving virus en níspero en España (Autora: A.B. Ruiz-García)
30/10/21 10:52 ..... Divulgacion
En prospecciones realizadas en áreas productoras de níspero se ha detectado por primera vez en España la presencia en este huésped leñoso del virus apple stem grooving virus con una incidencia de 6.59%.
La secuencia y caracterización genómica del primer aislado español de ASGV infectando níspero se realizó de la siguiente forma.
Se analizó por secuenciación masiva la muestra SL73.32 que dio como resultado 52.353.872 lecturas (con un promedio de tamaño de 134,34 nt). Se realizó el control de calidad con el recorte de adaptadores y de secuencias que no tenían la calidad suficiente. Las lecturas se mapearon contra el genoma de níspero para la sustracción del genoma del huésped y las 566.878 lecturas no relacionadas se sometieron a ensamblaje de novo, generando 519 contigs. Entre ellos, 7 contigs estaban relacionados, mediante análisis BLASTN/X, con el virus ASGV y tenían unos tamaños que oscilaron entre 1523 nt y 310 nt . El remapeo de las lecturas contra los contigs permitió la superposición entre algunos de los contigs y la recuperación de 3 secuencias parciales de 730, 1260 y 4291 nt que cubren el 96,7 % del genoma, faltando 98 nt en el extremo 5 prima; dos pequeñas regiones de codificación de 33 nt y 37 nt en el ORF1; y 48 nt en el extremo 3 prima, respecto a la secuencia de referencia (NC_001749). Para cubrir las dos brechas de ORF1, se realizaron RT-PCR y secuenciación de Sanger. La superposición entre las secuencias HTS parciales y las secuencias amplificadas por RT-PCR resultó en el ensamblaje de una secuencia de codificación ASGV de longitud casi completa de 6345 nt, SL73.32 (depositado en GenBank, número de acceso OK272504). SL73.32 mostró el mayor porcentaje de identidad de nucleótidos (95,1 %) con el aislado de cítricos FKSS2 (LC143387) de Japón y una similitud de nucleótidos del 83,42% con el aislado de níspero L3 de China (MK599422). Estos resultados confirman la aparición de ASGV en nísperos en España. El mapa de la organización genómica de SL73.32, cobertura promedio de HTS y similitud de proteínas de sus ORF con el aislado de ASGV más cercano (LC143387) se muestra en la Figura 1.

El estudio filogenético del aislado viral de níspero español con otros aislados en otros cultivos y con los aislados ASGV de cítricos (también denominados CTLV) se muestra en la Figura 2

La secuencia y caracterización genómica del primer aislado español de ASGV infectando níspero se realizó de la siguiente forma.
Se analizó por secuenciación masiva la muestra SL73.32 que dio como resultado 52.353.872 lecturas (con un promedio de tamaño de 134,34 nt). Se realizó el control de calidad con el recorte de adaptadores y de secuencias que no tenían la calidad suficiente. Las lecturas se mapearon contra el genoma de níspero para la sustracción del genoma del huésped y las 566.878 lecturas no relacionadas se sometieron a ensamblaje de novo, generando 519 contigs. Entre ellos, 7 contigs estaban relacionados, mediante análisis BLASTN/X, con el virus ASGV y tenían unos tamaños que oscilaron entre 1523 nt y 310 nt . El remapeo de las lecturas contra los contigs permitió la superposición entre algunos de los contigs y la recuperación de 3 secuencias parciales de 730, 1260 y 4291 nt que cubren el 96,7 % del genoma, faltando 98 nt en el extremo 5 prima; dos pequeñas regiones de codificación de 33 nt y 37 nt en el ORF1; y 48 nt en el extremo 3 prima, respecto a la secuencia de referencia (NC_001749). Para cubrir las dos brechas de ORF1, se realizaron RT-PCR y secuenciación de Sanger. La superposición entre las secuencias HTS parciales y las secuencias amplificadas por RT-PCR resultó en el ensamblaje de una secuencia de codificación ASGV de longitud casi completa de 6345 nt, SL73.32 (depositado en GenBank, número de acceso OK272504). SL73.32 mostró el mayor porcentaje de identidad de nucleótidos (95,1 %) con el aislado de cítricos FKSS2 (LC143387) de Japón y una similitud de nucleótidos del 83,42% con el aislado de níspero L3 de China (MK599422). Estos resultados confirman la aparición de ASGV en nísperos en España. El mapa de la organización genómica de SL73.32, cobertura promedio de HTS y similitud de proteínas de sus ORF con el aislado de ASGV más cercano (LC143387) se muestra en la Figura 1.

El estudio filogenético del aislado viral de níspero español con otros aislados en otros cultivos y con los aislados ASGV de cítricos (también denominados CTLV) se muestra en la Figura 2

Primera detección en España de PeSV en granado (Autora: A.B. Ruiz-García)
16/09/21 10:19 ..... Divulgacion
En nuestro laboratorio se analizaron muestras de hojas de granados que crecían en Alicante (España) y que mostraban manchas cloróticas a lo largo de las nervaduras. Para identificar con seguridad el agente causal se procedió a realizar un análisis mediante secuenciación masiva RNAseq (HTS, tecnología TrueSeq Illumina). Los datos se analizaron utilizando CLC Genomics Workbench 10.1.1. Se realizó el control de calidad y la sustracción del genoma del huésped. El ensamblado de novo de las 1887133 lecturas generó 10.946 contigs (> 200 nt), de los cuales 128 estaban relacionados con PeSV según el análisis BLAST (e-value < 10e–4). Estos contigs mostraron una alta variabilidad molecular lo que indicó una infección mixta por varios aislados de PeSV.
La extensión de los contig se realizó mediante el programa Geneious Prime y permitió recuperar una secuencia genómica de PeSV de 9926 nt casi completa (MZ361583, cobertura promedio 528x). Esta secuencia mostró una identidad de nucleótidos del 78,1 al 80,8 % en comparación con las secuencias genómicas de PeSV (MT680930-MT680935). No se detectó ningún otro virus en el análisis HTS.
La RT-PCR de la planta de granado original con los cebadores diseñados en este trabajo SPeSV-6F (5'-GGCTAGAAACGGTGGGATGA-3') y SPeSV-6R (5'-ACCACCTGGCTCATGGCGA-3') produjo un amplicón esperado de 165 nt, cuya secuencia fue confirmada por secuenciación de Sanger (100% de identidad de nt con la secuencia HTS).
Análisis posteriores sobre 64 árboles mostraron que 17 de ellos estaban infectados por PeSV. PeSV no se detectó en plantas asintomáticas. La mayor parte de árboles sintomáticos dieron positivo para PeSV aunque en alguno de ellos no se detectó por el método desarrollado lo que pone en evidencia la necesidad de mejorar los métodos de detección de este virus emergente en un cultivo de importancia en España como es el granado.

Para la primera descripción del granado como hospedado de PeSV y visualización con más detalle de la sintomatología producida por este virus se puede consultar la comunicación del 25 de noviembre de 2020.
La extensión de los contig se realizó mediante el programa Geneious Prime y permitió recuperar una secuencia genómica de PeSV de 9926 nt casi completa (MZ361583, cobertura promedio 528x). Esta secuencia mostró una identidad de nucleótidos del 78,1 al 80,8 % en comparación con las secuencias genómicas de PeSV (MT680930-MT680935). No se detectó ningún otro virus en el análisis HTS.
La RT-PCR de la planta de granado original con los cebadores diseñados en este trabajo SPeSV-6F (5'-GGCTAGAAACGGTGGGATGA-3') y SPeSV-6R (5'-ACCACCTGGCTCATGGCGA-3') produjo un amplicón esperado de 165 nt, cuya secuencia fue confirmada por secuenciación de Sanger (100% de identidad de nt con la secuencia HTS).
Análisis posteriores sobre 64 árboles mostraron que 17 de ellos estaban infectados por PeSV. PeSV no se detectó en plantas asintomáticas. La mayor parte de árboles sintomáticos dieron positivo para PeSV aunque en alguno de ellos no se detectó por el método desarrollado lo que pone en evidencia la necesidad de mejorar los métodos de detección de este virus emergente en un cultivo de importancia en España como es el granado.

Para la primera descripción del granado como hospedado de PeSV y visualización con más detalle de la sintomatología producida por este virus se puede consultar la comunicación del 25 de noviembre de 2020.
Primeras detecciones de plum bark necrosis stem pitting associated virus en cerezo dulce en España (Autora: A.B. Ruiz-García)
11/06/21 18:11 ..... Divulgacion
En prospecciones en un area de cultivo de cerezo se analizaron muestras para estudiar el estado sanitario del cerezo. En el marco de esta prospección, se analizó una muestra (P7) de un cerezo dulce (Prunus avium), del cultivar Planera, que mostraba enrojecimiento y manchas necróticas en las hojas y se analizó mediante secuenciación masiva (HTS) con la plataforma NextSeq 500. El análisis de datos se realizó utilizando el software CLC Genomics Workbench 10.1.1 y Geneious 9.1.8. Después del control de calidad realizado por el software CLC, la muestra P7 rindió 42.683.262 lecturas. El ensamblado de novo produjo un total de 6.270 contigs (tamaño promedio de 1.884 nt). Los contigs posteriormente se analizaron por BLASTN y BLASTX. Este análisis mostró que ocho contigs estaban relacionados con el virus plum bark necrosis stem pitting associated virus (PBNSPaV). Tabién se detectó la presencia del virus cherry virus A representado con la presencia de 11 contigs.
Se realizó un análisis adicional mediante el mapeo de las lecturas contra todas las secuencias completas de PBNSPaV disponibles en las bases de datos utilizando el software Geneious. Los mejores resultados de mapeo se obtuvieron usando el aislado Pair-2 (KC590345) de Francia como referencia, lo que permitió la recuperación de una secuencia de 14.199 nt, que representa el genoma casi completo del aislado P7 (número de acceso de GenBank MN228561).
La identidad de nucleótidos entre el aislado P7 y el Par-2 fue del 99,27 %. Para confirmar la presencia de PBNSPaV en la muestra P7, se realizó la amplificación de una región parcial del gen CP por RT-PCR utilizando los cebadores específicos PBN-CP-F y PBN-CP-R. El producto de PCR de 301 pb obtenido (MN240523) fue secuenciado por Sanger y confirmó con 100% de identidad de la secuencia P7 recuperada por HTS (excluyendo los cebadores usados para la amplificación).
Un total de 24 muestras recogidas de la misma zona de cultivo de cerezo se analizaron mediante RT-PCR utilizando los mismos cebadores. Siete de estas muestras dieron positivo para PBNSPaV, lo que confirma aún más la presencia del virus en cerezp dulce en España. Este es el primer informe de PBNSPaV infectando cerezo dulce en España, lo que contribuye a una mejor comprensión de la epidemiología y la distribución del rango de huéspedes de este patógeno.
Se realizó un análisis adicional mediante el mapeo de las lecturas contra todas las secuencias completas de PBNSPaV disponibles en las bases de datos utilizando el software Geneious. Los mejores resultados de mapeo se obtuvieron usando el aislado Pair-2 (KC590345) de Francia como referencia, lo que permitió la recuperación de una secuencia de 14.199 nt, que representa el genoma casi completo del aislado P7 (número de acceso de GenBank MN228561).
La identidad de nucleótidos entre el aislado P7 y el Par-2 fue del 99,27 %. Para confirmar la presencia de PBNSPaV en la muestra P7, se realizó la amplificación de una región parcial del gen CP por RT-PCR utilizando los cebadores específicos PBN-CP-F y PBN-CP-R. El producto de PCR de 301 pb obtenido (MN240523) fue secuenciado por Sanger y confirmó con 100% de identidad de la secuencia P7 recuperada por HTS (excluyendo los cebadores usados para la amplificación).
Un total de 24 muestras recogidas de la misma zona de cultivo de cerezo se analizaron mediante RT-PCR utilizando los mismos cebadores. Siete de estas muestras dieron positivo para PBNSPaV, lo que confirma aún más la presencia del virus en cerezp dulce en España. Este es el primer informe de PBNSPaV infectando cerezo dulce en España, lo que contribuye a una mejor comprensión de la epidemiología y la distribución del rango de huéspedes de este patógeno.
Actualización sobre las especies de virus que infectan vid (Autora: A.B. Ruiz-García)
26/04/21 20:05 ..... Divulgacion
Hasta la fecha, se han detectado 86 virus que infectan vides de todo el mundo. Algunos de estos virus se asocian con enfermedades económicamente perjudiciales y están asociados con los cuatro principales complejos de enfermedades conocidos como degeneración infecciosa, enrollado, madera rugosa y jaspeado.
A continuación una actualización de los virus que infectan vid:
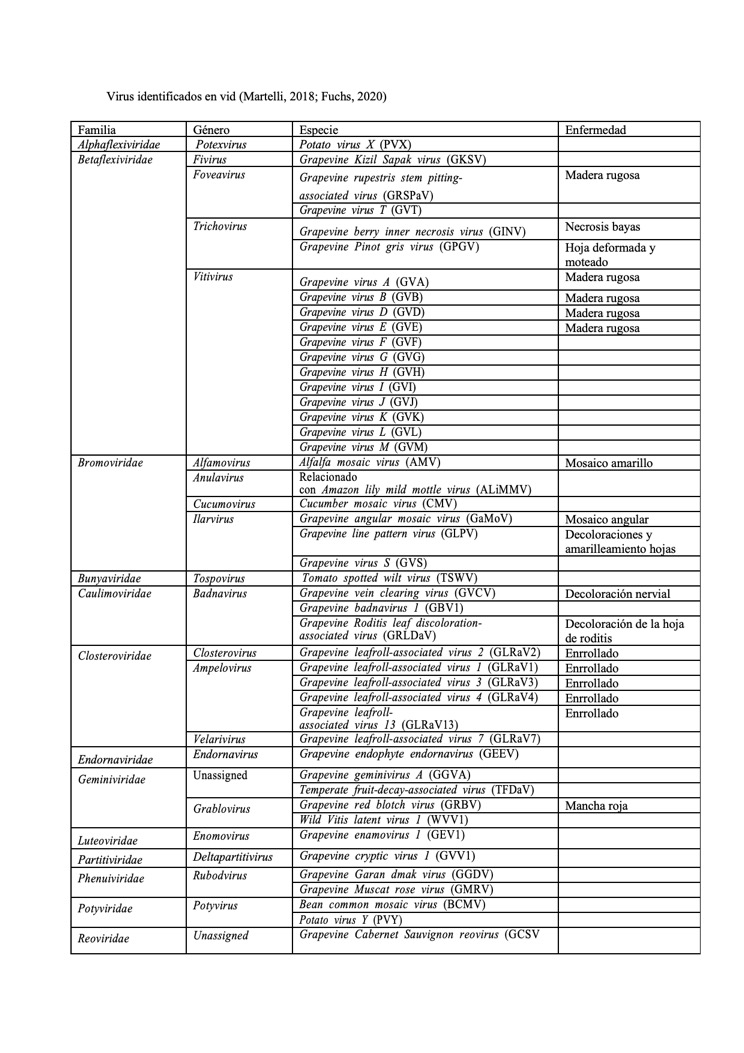
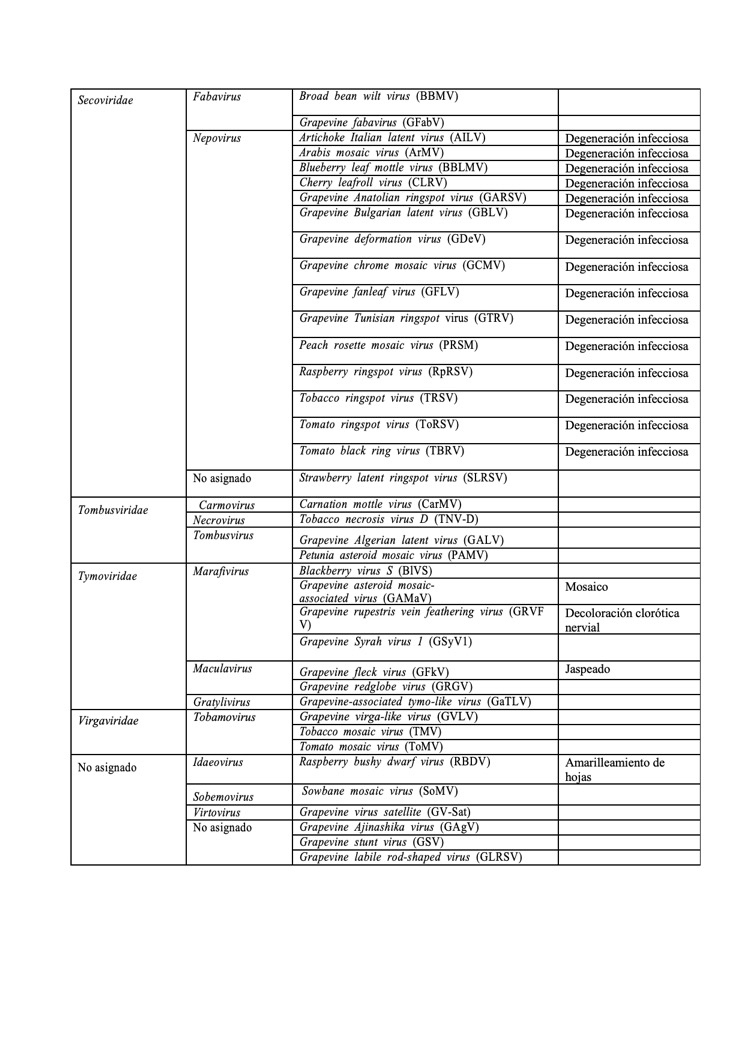
A continuación una actualización de los virus que infectan vid:


Recordando que el virus de los cítricos Citrus tatter feaf ha sido detectado por primera vez en Europa (Autora: A.B. Ruiz-García)
12/03/21 21:55 ..... Divulgacion
El virus de la hoja de cítricos Citrus tatter leaf virus (CTLV) es un capillovirus que pertenece a la familia Betaflexiviridae que se transmite de forma mecánica aunque no se han identificado todavía vectores.
Actualmente la ICTV lo considera como una cepa de un virus conocido de frutales, el Apple stem grooving virus (ASGV).
La mayoría de las variedades de cítricos comerciales infectadas son asintomáticas excepto cuando se propagan sobre patrones Poncirus trifoliata o Poncirus trifoliata x Citrus sinensis. Es entonces cuando los árboles presentan enanismo, hojas cloróticas, incompatibilidad en la unión del injerto lo que conlleva a al deterioro final del árbol. Esto supone un problema grave porque los patrones basados en Poncirus trifoliata y sus híbridos se utilizan ampliamente en todas las zonas productoras del mundo por su tolerancia al virus de la tristeza de los cítricos y a las especies de Phytophthora.
Este virus se describió por primera vez en California y posteriormente en Australia, Korea, Nigeria, Japan, Sudafrica, y China.
Sin embargo en el año 2019 (Turgut Alas et al., Saudi Journal of Biological Sciences 26, 995-998) detectaron este virus Chipre, siendo la primera detección del este virus en cítricos en Europa.
La sintomatología que los autores observaron fue la siguiente:

Distorsión de hojas, necrosis y síntomas cloróticos en hojas infectadas con CTLV (T. Alas et al. 2019)
Para la identificación del virus emplearon RT-PCR convencional empleando los iniciadores específicos:
TL1F: TGAAAACCTTTGCTGCCACCTCT
TL1R: TACTCTCCGAACCTGCCTCGAAA
que amplificaba un fragmento de 309 pb a una temperatura de annealing de 59ºC
Se trata pues de una detección muy a tener en cuenta puesto que este virus se ha considerado hasta la fecha como de cuarentena para la citricultura española.
Actualmente la ICTV lo considera como una cepa de un virus conocido de frutales, el Apple stem grooving virus (ASGV).
La mayoría de las variedades de cítricos comerciales infectadas son asintomáticas excepto cuando se propagan sobre patrones Poncirus trifoliata o Poncirus trifoliata x Citrus sinensis. Es entonces cuando los árboles presentan enanismo, hojas cloróticas, incompatibilidad en la unión del injerto lo que conlleva a al deterioro final del árbol. Esto supone un problema grave porque los patrones basados en Poncirus trifoliata y sus híbridos se utilizan ampliamente en todas las zonas productoras del mundo por su tolerancia al virus de la tristeza de los cítricos y a las especies de Phytophthora.
Este virus se describió por primera vez en California y posteriormente en Australia, Korea, Nigeria, Japan, Sudafrica, y China.
Sin embargo en el año 2019 (Turgut Alas et al., Saudi Journal of Biological Sciences 26, 995-998) detectaron este virus Chipre, siendo la primera detección del este virus en cítricos en Europa.
La sintomatología que los autores observaron fue la siguiente:

Distorsión de hojas, necrosis y síntomas cloróticos en hojas infectadas con CTLV (T. Alas et al. 2019)
Para la identificación del virus emplearon RT-PCR convencional empleando los iniciadores específicos:
TL1F: TGAAAACCTTTGCTGCCACCTCT
TL1R: TACTCTCCGAACCTGCCTCGAAA
que amplificaba un fragmento de 309 pb a una temperatura de annealing de 59ºC
Se trata pues de una detección muy a tener en cuenta puesto que este virus se ha considerado hasta la fecha como de cuarentena para la citricultura española.
¿Qué son los aislados Resistance-Breaking (RB) del virus de la tristeza de los cítricos? (Autora: A.B. Ruiz-García)
05/02/21 12:33 ..... Divulgacion
Tener controlada mediante patrones tolerantes la enfermedad de la tristeza de los cítricos causada por el virus CTV y que ha producido la muerte de más de 50 millones de árboles sólo en España, no quiere decir que debamos olvidar esta problemática.
Los virus evolucionan, se adaptan, mutan y recombinan para vencer resistencias de hospedadores y avanzar en su objetivo, su supervivencia. Y así han aparecido y se han descrito nuevos grupos o genotipos de CTV, como el caso de los aislados Resistance Breaking (RB) formados por un grupo de aislados con características genéticas y biológicas particulares, que se han dividido en dos genotipos, los del grupo 1 RB y los del grupo 2 RB.
Los aislados RB, a diferencia del resto de aislados del virus de la tristeza, pueden replicarse y moverse de forma sistémica en Poncirus trifoliata, que muestra resistencia a otros aislados de CTV.
Estos genotipos RB, se han descrito en Nueva Zelanda, República Dominicana, Puerto Rico, Sudáfrica, California, Brasil y Marruecos.
En España solo existen aislados poco agresivos de CTV, que producen tristeza cuando naranjos dulces, mandarinos, limas y pomelos se injertan sobre naranjo amargo, y se produce una incompatibilidad entre patrón y variedad, por lo que se mantiene como prioridad evitar la entrada de CTV no europeos que incluyen ahora los aislados RB para preservar nuestra citricultura.
Se han diseñado y validado los iniciadores específicos siguientes que permiten realizar PCRs convencionales. El protocolo de detección es el siguiente:
Muestreo: 5 brotes terminales (10 cm) alrededor de la copa del árbol
Purificación de ácidos nucleicos: RNeasy Plant kit o similar
Preparación del cóctel:
Para cada reacción de 25 microlitros (3 microlitros de RNA purificado)
H2O 10,7 microlitros
Tampón 5x Promega 5 microlitros
Cl2Mg 25 mM (uso 1,5 mM) 1,5 microlitros
dNTP (2,5 mM cada uno) uso 0,25 mM 2,5 microlitros
Iniciador F1 (25 microM) uso 1microM 1 microlitro
Inciador R1 (25 microM) uso 1microM 1 microlitro
Promega AMV (10U/microlitro) 0,1 microlitros
GoTaq G2 Promega (5 U/microlitro) 0,2 microlitros
Según el genotipo se seleccionan los iniciadores:
RB group1
Iniciador F1 AGT GGT GGA GAT TAC GTT G
Inciador R1 TAC ACG CGA CAA ATC GAG
RB group 2
Iniciador F1 CGG AAG GGA CTA CGT GGT
Iniciador R1 CGT TTG CAC GGG TTC AAT G
Condiciones del Termociclador
40 min -----------------42ºC
4 min ------------------- 94ºC
40 ciclos:
30 s ---------------------- 92ºC
30 s ---------------------- 60ºC
1 min -------------------- 72ºC
10 min ----------------- 72ºC
El granado nuevo hesped del virus passiflora edulis symptomless virus (Autora: A.B. Ruiz-García)
25/11/20 09:41 ..... Divulgacion
En uno de nuestros trabajos recientes (Caglayan, K.; Gazel, M.; Roumi, V.; Kocabag, H.D.; Tunç, B.; Reynard, J.S.; Ruiz-García, A.B.; Olmos, A. & Candresse, T. 2020. Identification of Pomegranate as a New Host of Passiflora Edulis Symptomless Virus (PeSV) and Analysis of PeSV Diversity. Agronomy 2020, 10, 1821) hemos identificado el virus pastaflora edulis symptomless virus (PeSV) en plantas de granado son sintomatología similar a la que produce el virus de la sharka. PeSV ha sido propuesto recientemente como un nuevo miembro de la familia Potiviridae, dentro del género Roymovirus. El genoma de las dos especies descritas de este género, rose yellow mosaic virus y PeSV muestran peculiaridades respecto a otros potyvirus, en particular, un sitio adicional de escisión de NIa-Pro en la proteína 6K2-NIa-VPg, la ausencia de motivos de transmisión de áfidos tanto en HC-Pro como en CP, así como la presencia de un supuesto motivo de transmisión de ácaros en la CP.

Figura 1: Síntomas producidos por la infección causada por PeSV en granado. Anillos cloróticos, clorosis nervial y anillos cloróticos a lo largo de las nervaduras centrales.
La estructura genómica de diversos aislados del virus se han obtenido y es común la confección de diferentes variantes virales en el mismo árbol.

Figura 2. Genoma del aislado (PeSV)-PS1-5.
Además en este estudio, se realizó microscopio electrónica y se observaron partículas similares a las de los potyvirus. Se emplearon plantas de granado sintomáticas para extraer los viriones. Los viriones que se observaron eran filamentos flexibles y con una longitud de entre 760 a 780 nm y de 11 a 20 nm de ancho, con simetría helicoidal.
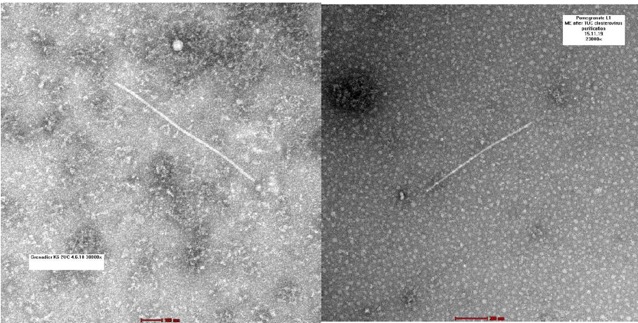
Figura 3. Microscopía electrónica empleando un microscopio de transmisión. La escala corresponde a 100nm en la fotografía de la izquierda y a 200 nm en la de la derecha.

Figura 1: Síntomas producidos por la infección causada por PeSV en granado. Anillos cloróticos, clorosis nervial y anillos cloróticos a lo largo de las nervaduras centrales.
La estructura genómica de diversos aislados del virus se han obtenido y es común la confección de diferentes variantes virales en el mismo árbol.

Figura 2. Genoma del aislado (PeSV)-PS1-5.
Además en este estudio, se realizó microscopio electrónica y se observaron partículas similares a las de los potyvirus. Se emplearon plantas de granado sintomáticas para extraer los viriones. Los viriones que se observaron eran filamentos flexibles y con una longitud de entre 760 a 780 nm y de 11 a 20 nm de ancho, con simetría helicoidal.

Figura 3. Microscopía electrónica empleando un microscopio de transmisión. La escala corresponde a 100nm en la fotografía de la izquierda y a 200 nm en la de la derecha.
Apple stem pitting virus, un virus de amplia distribución y que infecta diversas especies vegetales (Autora: A.B. Ruiz-García)
18/11/20 13:28 ..... Divulgacion
Apple stem pitting virus es un virus que está presente a nivel mundial y se ha descrito infectando manzano, peral, espino, membrillo y cerezo. A pesar de haber sido relacionado con problemas como epinastia, o punteaduras en madera en manzano, amarilleamiento de nervaduras o manchas necrosantes en pera y deformaciones en frutos de membrillo, de forma mucho más frecuente se ha relacionado a infecciones asintomáticas. Diversos estudios han visto la enorme variabilidad genética de este virus, y esta variabilidad se ha propuesta como la causa de su facilidad de adaptación a nuevos huéspedes.
En un estudio realizado en nuestro laboratorio en una prospección de níspero en la región de Segorbe, hemos detectado unas variantes capaces de infectar el níspero aunque sin producir sintomatología.
Para avanzar en el conocimiento y poder disponer de herramientas de diagnóstico de estas variantes de níspero, se ha secuenciado completamente un aislado y caracterizamos parcialmente 10 aislados más para ver filogenéticamente donde se situaban.
La estructura del aislado de níspero muestra que mantiene los cinco ORFs (RdRP, TGB1, TGB2, TGB3 y CP) como se muestra en la figura
El análisis filogenético del aislado completo de ASPV lo sitúa próximo a las variantes de peral en color verde.
El níspero se confirma como nuevo huésped de ASPV y se han diseñado iniciadores adaptados a la secuencia del aislado del níspero que ha permitido evaluar la prevalencia en Segorbe que ha resultado ser del 15%.
En un estudio realizado en nuestro laboratorio en una prospección de níspero en la región de Segorbe, hemos detectado unas variantes capaces de infectar el níspero aunque sin producir sintomatología.
Para avanzar en el conocimiento y poder disponer de herramientas de diagnóstico de estas variantes de níspero, se ha secuenciado completamente un aislado y caracterizamos parcialmente 10 aislados más para ver filogenéticamente donde se situaban.
La estructura del aislado de níspero muestra que mantiene los cinco ORFs (RdRP, TGB1, TGB2, TGB3 y CP) como se muestra en la figura
El análisis filogenético del aislado completo de ASPV lo sitúa próximo a las variantes de peral en color verde.
El níspero se confirma como nuevo huésped de ASPV y se han diseñado iniciadores adaptados a la secuencia del aislado del níspero que ha permitido evaluar la prevalencia en Segorbe que ha resultado ser del 15%.
Un misterio resuelto, el virus asociado al amarilleamiento de la hoja del olivo aporta datos clave para la creación de un nuevo género en la familia Closteroviridae, para el que se propone el nombre Olivavirus (Autora: A.B. Ruiz-García)
17/10/20 21:02 ..... Divulgacion
En la reciente publicación que tenemos (Ruiz-García, A.B.; Candresse, T.; Canales, C.; Morán, F.; Machado de Oliveira, C.; Bertolini, E.; Olmos, A. Molecular Characterization of the Complete Coding Sequence of Olive Leaf Yellowing-Associated Virus. Plants 2020, 9, 1272. https://doi.org/10.3390/plants9101272) se ha avanzado en el conocimiento del virus asociado al amarilleamiento de la hoja del olivo (OLYaV) y aporta información clave para la creación de un nuevo género en la familia Closteroviridae para el que se propone el nombre de Olivavirus.
El virus asociado al amarilleamiento de la hoja del olivo (OYLaV) se describió por primera vez en 1999, en Italia, en olivos de la variedad Biancolilla. Estos olivos mostraban decoloraciones del verde en las hojas, y tenían una coloración amarillo brillante. Sin embargo sólo se logró una caracterización parcial de un gen (HSP70) pero esta pequeña secuencia permitió que pudiera ser asignado a la familia Closteroviridae.
Estudios posteriores sólo avanzaron parcialmente en su conocimiento del genoma, ya que únicamente se logró conocer alrededor de 5000 nucleótidos. Con este conocimiento se diseñaron técnicas de diagnóstico y se averiguó que OLYaV era uno de los virus del olivo más extendidos en muchas zonas donde se cultivaba. Así, se descubrieron diferentes niveles de incidencia: California (EE. UU.) (93%), Italia (64-21%), Túnez (49%), Líbano (24%), Siria (15%), Grecia (5%) y Albania (2%).
Sin embargo OLYaV permanecía como una especie no asignada en la familia Closteroviridae porque se necesitaban más datos biológicos y moleculares para su clasificación.
En un estudio que recientemente hemos publicado, hemos descubierto la secuencia completa de OYLaV de 16700 nucleótidos, incluyendo todo su potencial de codificación.
En la figura se muestra la organización genómica de OLYaV. L-Pro: proteasa leader papain-like; Met-T: dominio metal transferasa; Helicase: dominio de la helicasa viral; RdRp: polimerasa RNA dependiente; Las flechas azules indican la posición de corte del proteasa.
La secuencia completa muestra un genoma con estructura típica de la familia Closteroviridae. Contiene 11 pautas de lectura abiertas (ORF,) entre ellos el complejo ORF1ab que codifica para los tres dominios conservados típicos, proteasa líder similar a la papaína, metiltransferasa y helicasa y además la RdRp, además de los genes de las proteínas HSP70 y HSP90.
OLYaV exhibe dos características genómicas originales. Primero, la ORF2 codifica una proteína similar a la taumatina y segundo, no codifica una proteína CPm. Curiosamente, estas dos características genómicas son compartidas por otras dos especies de Closteroviridae no asignadas, PVB and AV1. Además el análisis filogenético, fundamentalmente el estudio de los genes comunes en todas las especies de closterovirus (ORF1a, ORF1b (RdRp), HSP70, HSP90 y CP) ha abierto nuevas posibilidades en la clasificación taxonómica de OLYaV y del persimmon virus B (PVB) y el actidinia virus 1 (AV1), lo que sugiere de forma consistente que forman un nuevo género dentro de la familia Closteroviridae.
El virus asociado al amarilleamiento de la hoja del olivo (OYLaV) se describió por primera vez en 1999, en Italia, en olivos de la variedad Biancolilla. Estos olivos mostraban decoloraciones del verde en las hojas, y tenían una coloración amarillo brillante. Sin embargo sólo se logró una caracterización parcial de un gen (HSP70) pero esta pequeña secuencia permitió que pudiera ser asignado a la familia Closteroviridae.
Estudios posteriores sólo avanzaron parcialmente en su conocimiento del genoma, ya que únicamente se logró conocer alrededor de 5000 nucleótidos. Con este conocimiento se diseñaron técnicas de diagnóstico y se averiguó que OLYaV era uno de los virus del olivo más extendidos en muchas zonas donde se cultivaba. Así, se descubrieron diferentes niveles de incidencia: California (EE. UU.) (93%), Italia (64-21%), Túnez (49%), Líbano (24%), Siria (15%), Grecia (5%) y Albania (2%).
Sin embargo OLYaV permanecía como una especie no asignada en la familia Closteroviridae porque se necesitaban más datos biológicos y moleculares para su clasificación.
En un estudio que recientemente hemos publicado, hemos descubierto la secuencia completa de OYLaV de 16700 nucleótidos, incluyendo todo su potencial de codificación.
En la figura se muestra la organización genómica de OLYaV. L-Pro: proteasa leader papain-like; Met-T: dominio metal transferasa; Helicase: dominio de la helicasa viral; RdRp: polimerasa RNA dependiente; Las flechas azules indican la posición de corte del proteasa.
La secuencia completa muestra un genoma con estructura típica de la familia Closteroviridae. Contiene 11 pautas de lectura abiertas (ORF,) entre ellos el complejo ORF1ab que codifica para los tres dominios conservados típicos, proteasa líder similar a la papaína, metiltransferasa y helicasa y además la RdRp, además de los genes de las proteínas HSP70 y HSP90.
OLYaV exhibe dos características genómicas originales. Primero, la ORF2 codifica una proteína similar a la taumatina y segundo, no codifica una proteína CPm. Curiosamente, estas dos características genómicas son compartidas por otras dos especies de Closteroviridae no asignadas, PVB and AV1. Además el análisis filogenético, fundamentalmente el estudio de los genes comunes en todas las especies de closterovirus (ORF1a, ORF1b (RdRp), HSP70, HSP90 y CP) ha abierto nuevas posibilidades en la clasificación taxonómica de OLYaV y del persimmon virus B (PVB) y el actidinia virus 1 (AV1), lo que sugiere de forma consistente que forman un nuevo género dentro de la familia Closteroviridae.
Primera detección del Grapevine asteroid mosaic associated virus en viñas de Tempranillo y Macabeo en España (Autora: A.B. Ruiz-García)
27/09/20 22:13 ..... Divulgacion
El virus asociado al mosaico asteroide de la vid (GAMaV) pertenece al género Marafivirus de la familia Tymoviridae. GAMaV se encontró por primera vez infectando una vid en California y posteriormente se descubrió en Japón, Canadá, Uruguay, Francia, Hungría e Italia. En julio de 2019 una muestra de vid de la variedad Tempranillo (TS1), recogida durante uno de nuestros muestreos aleatorios en al D.O. Utiel-Requena, y que mostraba moteado clorótico y deformación en hojas, se analizó por secuenciación masiva (HTS). El análisis de esta muestra mostró que estaba infectada por diversos virus y viroides (grapevine rupestris stem pitting-associated virus, grapevine leafroll-associated virus 3, grapevine virus A, grapevine fleck virus, grapevine red globe virus, grapevine rupestris vein feathering virus y hop stunt viroid).
Además de estos se descubrió la presencia del virus asteroide de la vid y la secuencia completa se recuperó siendo de 6692 nucletótidos. El análisis filogenético mostró que el aislado español se situaba en un clado diferente a los que previamente estaban descritos, como se muestra en la figura.
Basándonos en la secuencia del aislado TS1 diseñamos dos iniciadores - GAMaV-6010F, 5’CCCTCCTCCTAGCGACGACC3’ y GAMaV- 6426R, 5’GGGTTGAGACGGCGGAGATC3’ - que amplifican un fragmento de 417 nt en la región de la cáspida y que ha permitido detectar los aislados españoles con fiabilidad, revelando una incidencia en el area estudiada de casi el 10%, puesto que los análisis con los iniciadores descritos por otros autores no detectaban los españoles. Estas han sido las primeras detecciones en España de este virus.
Además de estos se descubrió la presencia del virus asteroide de la vid y la secuencia completa se recuperó siendo de 6692 nucletótidos. El análisis filogenético mostró que el aislado español se situaba en un clado diferente a los que previamente estaban descritos, como se muestra en la figura.
Basándonos en la secuencia del aislado TS1 diseñamos dos iniciadores - GAMaV-6010F, 5’CCCTCCTCCTAGCGACGACC3’ y GAMaV- 6426R, 5’GGGTTGAGACGGCGGAGATC3’ - que amplifican un fragmento de 417 nt en la región de la cáspida y que ha permitido detectar los aislados españoles con fiabilidad, revelando una incidencia en el area estudiada de casi el 10%, puesto que los análisis con los iniciadores descritos por otros autores no detectaban los españoles. Estas han sido las primeras detecciones en España de este virus.
 orcid.org/0000-0001-8406-7963
orcid.org/0000-0001-8406-7963