February 2020
Un virus emergente en vid, Grapevine pinot gris virus (Autora: A.B. Ruiz-García)
01/02/20 16:03 ..... Divulgacion
Hacia el año 2004 una nueva enfermedad en vid apareció en Italia en Trentino, que producía retraso y reducción del crecimiento general de la planta, pérdida de vigor de las cepas, enanismo en sarmientos y que presentaba malformaciones en hojas y moteados cloróticos. La aplicación de la tecnología de la secuenciación masiva permitió a un grupo de investigación italiano en el año 2012 la identificación de un nuevo virus del género Trichovirus, de la familia Betaflexiviridae, asociado a cepas que presentaban síntomas de la enfermedad. El genoma del virus consiste en una molécula de ARN de polaridad positiva de entre 7223 y 7275 nucleótidos excluyendo la cola poli A en el extremo 3’ del virus. El genoma contiene tres marcos de lectura abiertos (ORF) solapantes y dos regiones no traducidas, una en el extremo 5’ y otra en el extremo 3’. El primer ORF codifica una metiltransferasa, una helicasa y la ARN polimerasa dependiente de ARN, el segundo ORF codifica la proteína de movimiento, y el tercer ORF la proteína de la cáspida.
Desde su primera descripción en Italia, el virus se ha descrito en otros países como Grecia, República Checa, Eslovaquia, Eslovenia, Francia, Alemania, Portugal, Corea del Sur, China, Canadá, EEUU, Turquía, Brasil, Croacia, Rumanía, Ucrania o Australia.
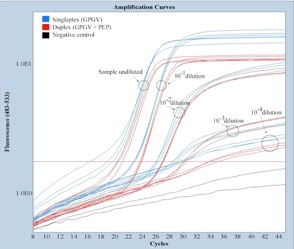
En España nuestro grupo de investigación describió por primera su presencia en el país en el año 2017, infectando variedades de Garnacha, Bobal y Tempranillo. Con el objetivo de comprender y mejorar la epidemiología de la enfermedad y desarrollar estrategias de control hemos desarrollado una RT-PCR a tiempo real que permite la detección y cuantificación del virus, con un limite de 70 copias del virus en material vegetal. También se ha aplicado a la detección en un posible vector Colomerus vitis.

Además hemos descubierto un nuevo polimorfismo en el genoma del virus que hace que la proteína de movimiento sea más pequeña.
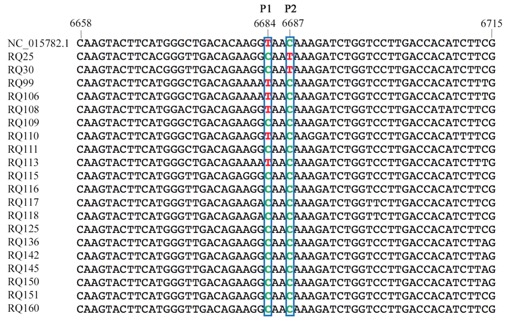
Un estudio filogenético basado en esta región genómica muestra una alta variaibilidad entre los aislados españoles, que se localizan en diversos grupos o “clusters” independientemente de tener o no polimorfismos en la proteína de movimiento (nuevo polimorfismo, círculo rojo completo; polimorfismo previamente descrito por un grupo italiano, círculo con sólo la circunferencia roja)
El desarrollo de esta metodología de detección y el avance en la diversidad del virus, permitirá mejorar herramientas de control y diagnóstico del virus.
Desde su primera descripción en Italia, el virus se ha descrito en otros países como Grecia, República Checa, Eslovaquia, Eslovenia, Francia, Alemania, Portugal, Corea del Sur, China, Canadá, EEUU, Turquía, Brasil, Croacia, Rumanía, Ucrania o Australia.
En España nuestro grupo de investigación describió por primera su presencia en el país en el año 2017, infectando variedades de Garnacha, Bobal y Tempranillo. Con el objetivo de comprender y mejorar la epidemiología de la enfermedad y desarrollar estrategias de control hemos desarrollado una RT-PCR a tiempo real que permite la detección y cuantificación del virus, con un limite de 70 copias del virus en material vegetal. También se ha aplicado a la detección en un posible vector Colomerus vitis.


Además hemos descubierto un nuevo polimorfismo en el genoma del virus que hace que la proteína de movimiento sea más pequeña.

Un estudio filogenético basado en esta región genómica muestra una alta variaibilidad entre los aislados españoles, que se localizan en diversos grupos o “clusters” independientemente de tener o no polimorfismos en la proteína de movimiento (nuevo polimorfismo, círculo rojo completo; polimorfismo previamente descrito por un grupo italiano, círculo con sólo la circunferencia roja)
El desarrollo de esta metodología de detección y el avance en la diversidad del virus, permitirá mejorar herramientas de control y diagnóstico del virus.
Comments
 orcid.org/0000-0001-8406-7963
orcid.org/0000-0001-8406-7963