olivavirus
Un misterio resuelto, el virus asociado al amarilleamiento de la hoja del olivo aporta datos clave para la creación de un nuevo género en la familia Closteroviridae, para el que se propone el nombre Olivavirus (Autora: A.B. Ruiz-García)
17/10/20 21:02 ..... Divulgacion
En la reciente publicación que tenemos (Ruiz-García, A.B.; Candresse, T.; Canales, C.; Morán, F.; Machado de Oliveira, C.; Bertolini, E.; Olmos, A. Molecular Characterization of the Complete Coding Sequence of Olive Leaf Yellowing-Associated Virus. Plants 2020, 9, 1272. https://doi.org/10.3390/plants9101272) se ha avanzado en el conocimiento del virus asociado al amarilleamiento de la hoja del olivo (OLYaV) y aporta información clave para la creación de un nuevo género en la familia Closteroviridae para el que se propone el nombre de Olivavirus.
El virus asociado al amarilleamiento de la hoja del olivo (OYLaV) se describió por primera vez en 1999, en Italia, en olivos de la variedad Biancolilla. Estos olivos mostraban decoloraciones del verde en las hojas, y tenían una coloración amarillo brillante. Sin embargo sólo se logró una caracterización parcial de un gen (HSP70) pero esta pequeña secuencia permitió que pudiera ser asignado a la familia Closteroviridae.
Estudios posteriores sólo avanzaron parcialmente en su conocimiento del genoma, ya que únicamente se logró conocer alrededor de 5000 nucleótidos. Con este conocimiento se diseñaron técnicas de diagnóstico y se averiguó que OLYaV era uno de los virus del olivo más extendidos en muchas zonas donde se cultivaba. Así, se descubrieron diferentes niveles de incidencia: California (EE. UU.) (93%), Italia (64-21%), Túnez (49%), Líbano (24%), Siria (15%), Grecia (5%) y Albania (2%).
Sin embargo OLYaV permanecía como una especie no asignada en la familia Closteroviridae porque se necesitaban más datos biológicos y moleculares para su clasificación.
En un estudio que recientemente hemos publicado, hemos descubierto la secuencia completa de OYLaV de 16700 nucleótidos, incluyendo todo su potencial de codificación.
En la figura se muestra la organización genómica de OLYaV. L-Pro: proteasa leader papain-like; Met-T: dominio metal transferasa; Helicase: dominio de la helicasa viral; RdRp: polimerasa RNA dependiente; Las flechas azules indican la posición de corte del proteasa.
La secuencia completa muestra un genoma con estructura típica de la familia Closteroviridae. Contiene 11 pautas de lectura abiertas (ORF,) entre ellos el complejo ORF1ab que codifica para los tres dominios conservados típicos, proteasa líder similar a la papaína, metiltransferasa y helicasa y además la RdRp, además de los genes de las proteínas HSP70 y HSP90.
OLYaV exhibe dos características genómicas originales. Primero, la ORF2 codifica una proteína similar a la taumatina y segundo, no codifica una proteína CPm. Curiosamente, estas dos características genómicas son compartidas por otras dos especies de Closteroviridae no asignadas, PVB and AV1. Además el análisis filogenético, fundamentalmente el estudio de los genes comunes en todas las especies de closterovirus (ORF1a, ORF1b (RdRp), HSP70, HSP90 y CP) ha abierto nuevas posibilidades en la clasificación taxonómica de OLYaV y del persimmon virus B (PVB) y el actidinia virus 1 (AV1), lo que sugiere de forma consistente que forman un nuevo género dentro de la familia Closteroviridae.
El virus asociado al amarilleamiento de la hoja del olivo (OYLaV) se describió por primera vez en 1999, en Italia, en olivos de la variedad Biancolilla. Estos olivos mostraban decoloraciones del verde en las hojas, y tenían una coloración amarillo brillante. Sin embargo sólo se logró una caracterización parcial de un gen (HSP70) pero esta pequeña secuencia permitió que pudiera ser asignado a la familia Closteroviridae.
Estudios posteriores sólo avanzaron parcialmente en su conocimiento del genoma, ya que únicamente se logró conocer alrededor de 5000 nucleótidos. Con este conocimiento se diseñaron técnicas de diagnóstico y se averiguó que OLYaV era uno de los virus del olivo más extendidos en muchas zonas donde se cultivaba. Así, se descubrieron diferentes niveles de incidencia: California (EE. UU.) (93%), Italia (64-21%), Túnez (49%), Líbano (24%), Siria (15%), Grecia (5%) y Albania (2%).
Sin embargo OLYaV permanecía como una especie no asignada en la familia Closteroviridae porque se necesitaban más datos biológicos y moleculares para su clasificación.
En un estudio que recientemente hemos publicado, hemos descubierto la secuencia completa de OYLaV de 16700 nucleótidos, incluyendo todo su potencial de codificación.
En la figura se muestra la organización genómica de OLYaV. L-Pro: proteasa leader papain-like; Met-T: dominio metal transferasa; Helicase: dominio de la helicasa viral; RdRp: polimerasa RNA dependiente; Las flechas azules indican la posición de corte del proteasa.
La secuencia completa muestra un genoma con estructura típica de la familia Closteroviridae. Contiene 11 pautas de lectura abiertas (ORF,) entre ellos el complejo ORF1ab que codifica para los tres dominios conservados típicos, proteasa líder similar a la papaína, metiltransferasa y helicasa y además la RdRp, además de los genes de las proteínas HSP70 y HSP90.
OLYaV exhibe dos características genómicas originales. Primero, la ORF2 codifica una proteína similar a la taumatina y segundo, no codifica una proteína CPm. Curiosamente, estas dos características genómicas son compartidas por otras dos especies de Closteroviridae no asignadas, PVB and AV1. Además el análisis filogenético, fundamentalmente el estudio de los genes comunes en todas las especies de closterovirus (ORF1a, ORF1b (RdRp), HSP70, HSP90 y CP) ha abierto nuevas posibilidades en la clasificación taxonómica de OLYaV y del persimmon virus B (PVB) y el actidinia virus 1 (AV1), lo que sugiere de forma consistente que forman un nuevo género dentro de la familia Closteroviridae.
Comments
Estudios en el viroma del olivo en España, el olivavirus OLYaV (Autora: A.B. Ruiz-García)
12/01/22 13:22 ..... Divulgacion
En un reciente trabajo el uso de secuenciación masiva (HTS) para el análisis de olivos españoles que mostraban decoloración de hojas y amarilleamiento, la defoliación y/o el declive ha proporcionado nuevos conocimientos sobre los virus de olivo presentes en España y ha abierto debates sobre las ventajas e inconvenientes de estas tecnologías con fines diagnósticos (Ruiz-García AB, Canales C, Morán F, Ruiz-Torres M, Herrera-Mármol M, Olmos A. Characterization of Spanish Olive Virome by High Throughput Sequencing Opens New Insights and Uncertainties. Viruses. 2021 Nov 6;13:2233). En este estudio describimos por primera vez en el olivar español la presencia del virus asociado al amarillamiento de la hoja del olivo (OLYaV), un virus cuya secuencia completa fue obtenida por nuestro grupo previamente y que ofrece información para la creación de un nuevo género (Olivavirus) en la familia Closteroviridae (véase la comunicación del 17/10/2020).
En la figura 1 se muestra la sintomatología observada en campo en árboles de olivo.

Figura 1. Síntomas de defoliación, muerte y amarilleamiento de hojas causados por OLYaV.
La comparación entre las secuencias nucleotídica/proteínica del nuevo aislado español de OLYaV y las secuencias nucleotídica/proteínica del aislado brasileño previamente secuenciado por nuestro equipo también, se muestra en la figura 2.
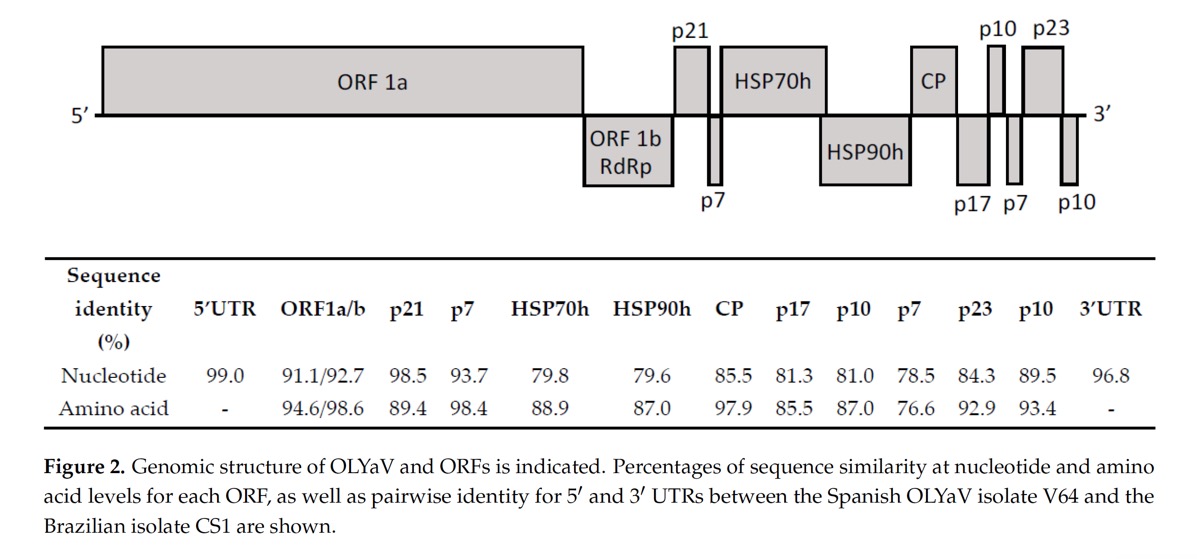
Como se puede observar las regiones más óptimas para la detección del virus deberán basarse en la región 5’UTR, p21 y/o 3’UTR y no la HSP70 que es la que actualmente se emplea y tiene un bajo porcentaje de homología (79.8%) lo que implica la presencia de falsos negativos porque la técnica puede no cubrir la variabilidad del virus.
Además se ha obtenido la segunda secuencia completa de OLYaV. Este virus también se ha detectado en un posible vector, el psílido Euphyllura olivina. Además, se ha confirmado en España también la presencia de Olea europaea geminivirus (OEGV), recientemente informado en Italia, y se ha obtenido la secuencia completa de dos aislados por secuenciación HTS y por secuenciación Sanger.
En la figura 1 se muestra la sintomatología observada en campo en árboles de olivo.

Figura 1. Síntomas de defoliación, muerte y amarilleamiento de hojas causados por OLYaV.
La comparación entre las secuencias nucleotídica/proteínica del nuevo aislado español de OLYaV y las secuencias nucleotídica/proteínica del aislado brasileño previamente secuenciado por nuestro equipo también, se muestra en la figura 2.

Como se puede observar las regiones más óptimas para la detección del virus deberán basarse en la región 5’UTR, p21 y/o 3’UTR y no la HSP70 que es la que actualmente se emplea y tiene un bajo porcentaje de homología (79.8%) lo que implica la presencia de falsos negativos porque la técnica puede no cubrir la variabilidad del virus.
Además se ha obtenido la segunda secuencia completa de OLYaV. Este virus también se ha detectado en un posible vector, el psílido Euphyllura olivina. Además, se ha confirmado en España también la presencia de Olea europaea geminivirus (OEGV), recientemente informado en Italia, y se ha obtenido la secuencia completa de dos aislados por secuenciación HTS y por secuenciación Sanger.
 orcid.org/0000-0001-8406-7963
orcid.org/0000-0001-8406-7963