2021
El níspero como nuevo huesped del viroide del manzano de cabeza de martillo (Autora: A.B. Ruiz-García)
16/12/21 12:32 ..... Divulgacion
En prospecciones de níspero en zonas productoras se detectó por primera vez como huésped alternativo un viroide que había sido descrito infectando manzano. El viroide del manzano de cabeza de martillo (AHVd). El viroide se identificó en una muestra de níspero la SL73.6 que se analizó por secuenciacón masiva. Entre los contigs relacionados con virus/viroides ensamblados a partir de datos SL73.6 HTS, se encontraron dos contigs de 489 y 376 nt relacionados con el viroide AHVd. El análisis bioinformático de estos contigs permitió la recuperación de un genoma circular completo de 376 nt cubierto por 7014 lecturas (cobertura promedio 2515.5x), el aislado AHVd-SL73.6 (depositado en GenBank, número de acceso OK272503). Para confirmar la presencia de AHVd en esta muestra, el genoma completo se secuenció mediante Sanger tras la amplificada por RT-PCR utilizando cebadores adyacentes de opuesta polaridad. Se obtuvo un producto de 376 pb, que se clonó y al secuenciarlo mediante Sanger, se confirmó la secuencia obtenida por HTS al 100%. Estos resultados muestran por primera vez la presencia de AHVd infectando al níspero, identificando así a este cultivo como un nuevo huésped de esta especie de viroide. El aislado de níspero español AHVd mostró una alta identidad de nucleótidos (93,97%) con el aislado de referencia (NC_028132, KR605506) de China, aunque el genoma AHVd-SL73.6 fue más corto (376 nt frente a 434 nt) debido a una eliminación de 56 nt en la posición genómica 55-110 y una deleción de 2 nt en la posición genómica 419-420 con respecto a la secuencia de referencia. Sin embargo, la estructura secundaria predicha para AHVd-SL73.6 mostró un plegamiento similar a los descritos previamente, resultando en una conformación característica de Pelamoviroides compuestos por un dominio en forma de barilla que contiene los nucleótidos que forman la estructura de cabeza de martillo y un dominio multiramificado.
En la siguiente figura se observa la estructura secundaria del viroide aislado de níspero.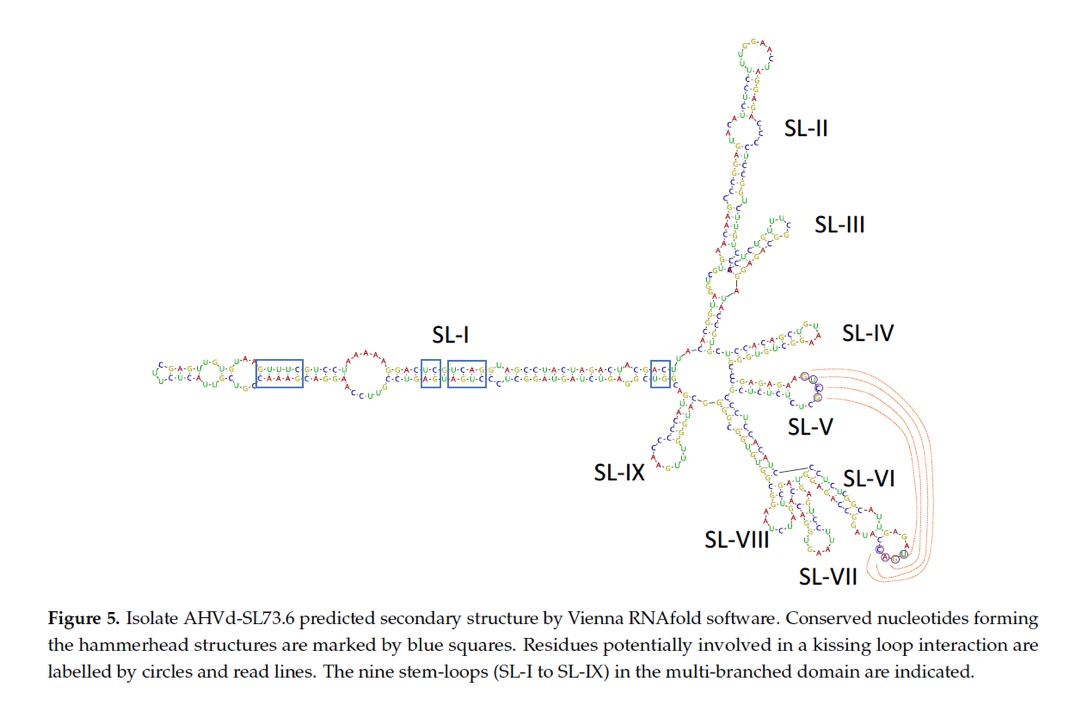
En la siguiente figura se observa la estructura secundaria del viroide aislado de níspero.

Comments
Primeras detecciones de la falsa sharka en níspero en España (Autora: A.B. Ruiz-García)
03/11/21 11:26 ..... Divulgacion
Continuando con el níspero y avanzar en el conocimiento de su estado sanitario en España, en prospecciones realizadas en áreas productoras de níspero se ha detectado por primera la presencia del virus de la falsa sharka, apple clorotic leaf spot virus (ACLSV) en níspero con una alta incidencia del 31.76%.
La secuencia y caracterización genómica del primer aislado español de ACLSV infectando níspero se realizó de la siguiente forma.
El análisis por secuenciación masiva (HTS) realizado en el ARN total extraído de la muestra SL73.6 rindió 31.867.726 lecturas d con un tamaño promedio de 134,8 nt. Se realizó la sustracción del genoma del níspero, tras el control de calidad de las lecturas y eliminar los adaptadores, lo que resultó en 1.698.300 lecturas que se utilizaron para el ensamblado de novo que dio lugar a 13.797 contigs. Se anotaron los contigs relacionados con virus
y viroides mediante BLASTN/X. Este análisis mostró 11 contigs de tamaños entre
6511 y 281 nt relacionados con ACLSV, confirmando la presencia de ACLSV en la muestra. La extensión de los contigs realizando el mapeo de las lecturas contra los contigs permitió la recuperación de un genoma casi completo de 7533 nt, cubierto por 44 039 lecturas (cobertura promedio 857,2x), aislado llamado SL73.6 y depositado en GenBank, número de acceso OK272502. Este aislado mostró el mayor porcentaje de identidad a nivel nueclotídico (83,7%) con el aislado alemán 38/85-B (KX579123) de manzano. El mapa de la organización del genoma SL73.6, la cobertura promedio de HTS y la similitud de proteínas de sus ORF con el aislado de ACLSV más cercano (KX579123) se muestran en la Figura1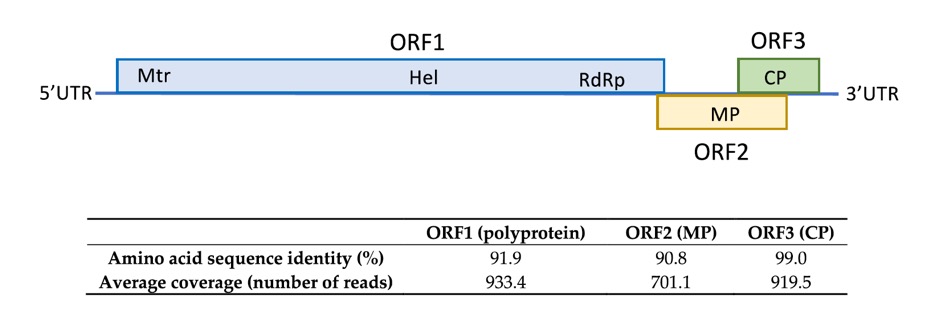
Figura 1. Similaridad aminoacídica del aislado SL73.6 con los ORFs del aislado de ACLSV más próximo (KX579123). Se muestra la cobertura por HTS.
En la siguiente figura se muestra la relación filogenética del aislado de ACLSV de níspero con otros aislados de ACLSV de otros orígenes y de otros hospedados.

La secuencia y caracterización genómica del primer aislado español de ACLSV infectando níspero se realizó de la siguiente forma.
El análisis por secuenciación masiva (HTS) realizado en el ARN total extraído de la muestra SL73.6 rindió 31.867.726 lecturas d con un tamaño promedio de 134,8 nt. Se realizó la sustracción del genoma del níspero, tras el control de calidad de las lecturas y eliminar los adaptadores, lo que resultó en 1.698.300 lecturas que se utilizaron para el ensamblado de novo que dio lugar a 13.797 contigs. Se anotaron los contigs relacionados con virus
y viroides mediante BLASTN/X. Este análisis mostró 11 contigs de tamaños entre
6511 y 281 nt relacionados con ACLSV, confirmando la presencia de ACLSV en la muestra. La extensión de los contigs realizando el mapeo de las lecturas contra los contigs permitió la recuperación de un genoma casi completo de 7533 nt, cubierto por 44 039 lecturas (cobertura promedio 857,2x), aislado llamado SL73.6 y depositado en GenBank, número de acceso OK272502. Este aislado mostró el mayor porcentaje de identidad a nivel nueclotídico (83,7%) con el aislado alemán 38/85-B (KX579123) de manzano. El mapa de la organización del genoma SL73.6, la cobertura promedio de HTS y la similitud de proteínas de sus ORF con el aislado de ACLSV más cercano (KX579123) se muestran en la Figura1

Figura 1. Similaridad aminoacídica del aislado SL73.6 con los ORFs del aislado de ACLSV más próximo (KX579123). Se muestra la cobertura por HTS.
En la siguiente figura se muestra la relación filogenética del aislado de ACLSV de níspero con otros aislados de ACLSV de otros orígenes y de otros hospedados.

Primeras detecciones de apple stem grooving virus en níspero en España (Autora: A.B. Ruiz-García)
30/10/21 10:52 ..... Divulgacion
En prospecciones realizadas en áreas productoras de níspero se ha detectado por primera vez en España la presencia en este huésped leñoso del virus apple stem grooving virus con una incidencia de 6.59%.
La secuencia y caracterización genómica del primer aislado español de ASGV infectando níspero se realizó de la siguiente forma.
Se analizó por secuenciación masiva la muestra SL73.32 que dio como resultado 52.353.872 lecturas (con un promedio de tamaño de 134,34 nt). Se realizó el control de calidad con el recorte de adaptadores y de secuencias que no tenían la calidad suficiente. Las lecturas se mapearon contra el genoma de níspero para la sustracción del genoma del huésped y las 566.878 lecturas no relacionadas se sometieron a ensamblaje de novo, generando 519 contigs. Entre ellos, 7 contigs estaban relacionados, mediante análisis BLASTN/X, con el virus ASGV y tenían unos tamaños que oscilaron entre 1523 nt y 310 nt . El remapeo de las lecturas contra los contigs permitió la superposición entre algunos de los contigs y la recuperación de 3 secuencias parciales de 730, 1260 y 4291 nt que cubren el 96,7 % del genoma, faltando 98 nt en el extremo 5 prima; dos pequeñas regiones de codificación de 33 nt y 37 nt en el ORF1; y 48 nt en el extremo 3 prima, respecto a la secuencia de referencia (NC_001749). Para cubrir las dos brechas de ORF1, se realizaron RT-PCR y secuenciación de Sanger. La superposición entre las secuencias HTS parciales y las secuencias amplificadas por RT-PCR resultó en el ensamblaje de una secuencia de codificación ASGV de longitud casi completa de 6345 nt, SL73.32 (depositado en GenBank, número de acceso OK272504). SL73.32 mostró el mayor porcentaje de identidad de nucleótidos (95,1 %) con el aislado de cítricos FKSS2 (LC143387) de Japón y una similitud de nucleótidos del 83,42% con el aislado de níspero L3 de China (MK599422). Estos resultados confirman la aparición de ASGV en nísperos en España. El mapa de la organización genómica de SL73.32, cobertura promedio de HTS y similitud de proteínas de sus ORF con el aislado de ASGV más cercano (LC143387) se muestra en la Figura 1.

El estudio filogenético del aislado viral de níspero español con otros aislados en otros cultivos y con los aislados ASGV de cítricos (también denominados CTLV) se muestra en la Figura 2

La secuencia y caracterización genómica del primer aislado español de ASGV infectando níspero se realizó de la siguiente forma.
Se analizó por secuenciación masiva la muestra SL73.32 que dio como resultado 52.353.872 lecturas (con un promedio de tamaño de 134,34 nt). Se realizó el control de calidad con el recorte de adaptadores y de secuencias que no tenían la calidad suficiente. Las lecturas se mapearon contra el genoma de níspero para la sustracción del genoma del huésped y las 566.878 lecturas no relacionadas se sometieron a ensamblaje de novo, generando 519 contigs. Entre ellos, 7 contigs estaban relacionados, mediante análisis BLASTN/X, con el virus ASGV y tenían unos tamaños que oscilaron entre 1523 nt y 310 nt . El remapeo de las lecturas contra los contigs permitió la superposición entre algunos de los contigs y la recuperación de 3 secuencias parciales de 730, 1260 y 4291 nt que cubren el 96,7 % del genoma, faltando 98 nt en el extremo 5 prima; dos pequeñas regiones de codificación de 33 nt y 37 nt en el ORF1; y 48 nt en el extremo 3 prima, respecto a la secuencia de referencia (NC_001749). Para cubrir las dos brechas de ORF1, se realizaron RT-PCR y secuenciación de Sanger. La superposición entre las secuencias HTS parciales y las secuencias amplificadas por RT-PCR resultó en el ensamblaje de una secuencia de codificación ASGV de longitud casi completa de 6345 nt, SL73.32 (depositado en GenBank, número de acceso OK272504). SL73.32 mostró el mayor porcentaje de identidad de nucleótidos (95,1 %) con el aislado de cítricos FKSS2 (LC143387) de Japón y una similitud de nucleótidos del 83,42% con el aislado de níspero L3 de China (MK599422). Estos resultados confirman la aparición de ASGV en nísperos en España. El mapa de la organización genómica de SL73.32, cobertura promedio de HTS y similitud de proteínas de sus ORF con el aislado de ASGV más cercano (LC143387) se muestra en la Figura 1.

El estudio filogenético del aislado viral de níspero español con otros aislados en otros cultivos y con los aislados ASGV de cítricos (también denominados CTLV) se muestra en la Figura 2

Primera detección en España de PeSV en granado (Autora: A.B. Ruiz-García)
16/09/21 10:19 ..... Divulgacion
En nuestro laboratorio se analizaron muestras de hojas de granados que crecían en Alicante (España) y que mostraban manchas cloróticas a lo largo de las nervaduras. Para identificar con seguridad el agente causal se procedió a realizar un análisis mediante secuenciación masiva RNAseq (HTS, tecnología TrueSeq Illumina). Los datos se analizaron utilizando CLC Genomics Workbench 10.1.1. Se realizó el control de calidad y la sustracción del genoma del huésped. El ensamblado de novo de las 1887133 lecturas generó 10.946 contigs (> 200 nt), de los cuales 128 estaban relacionados con PeSV según el análisis BLAST (e-value < 10e–4). Estos contigs mostraron una alta variabilidad molecular lo que indicó una infección mixta por varios aislados de PeSV.
La extensión de los contig se realizó mediante el programa Geneious Prime y permitió recuperar una secuencia genómica de PeSV de 9926 nt casi completa (MZ361583, cobertura promedio 528x). Esta secuencia mostró una identidad de nucleótidos del 78,1 al 80,8 % en comparación con las secuencias genómicas de PeSV (MT680930-MT680935). No se detectó ningún otro virus en el análisis HTS.
La RT-PCR de la planta de granado original con los cebadores diseñados en este trabajo SPeSV-6F (5'-GGCTAGAAACGGTGGGATGA-3') y SPeSV-6R (5'-ACCACCTGGCTCATGGCGA-3') produjo un amplicón esperado de 165 nt, cuya secuencia fue confirmada por secuenciación de Sanger (100% de identidad de nt con la secuencia HTS).
Análisis posteriores sobre 64 árboles mostraron que 17 de ellos estaban infectados por PeSV. PeSV no se detectó en plantas asintomáticas. La mayor parte de árboles sintomáticos dieron positivo para PeSV aunque en alguno de ellos no se detectó por el método desarrollado lo que pone en evidencia la necesidad de mejorar los métodos de detección de este virus emergente en un cultivo de importancia en España como es el granado.

Para la primera descripción del granado como hospedado de PeSV y visualización con más detalle de la sintomatología producida por este virus se puede consultar la comunicación del 25 de noviembre de 2020.
La extensión de los contig se realizó mediante el programa Geneious Prime y permitió recuperar una secuencia genómica de PeSV de 9926 nt casi completa (MZ361583, cobertura promedio 528x). Esta secuencia mostró una identidad de nucleótidos del 78,1 al 80,8 % en comparación con las secuencias genómicas de PeSV (MT680930-MT680935). No se detectó ningún otro virus en el análisis HTS.
La RT-PCR de la planta de granado original con los cebadores diseñados en este trabajo SPeSV-6F (5'-GGCTAGAAACGGTGGGATGA-3') y SPeSV-6R (5'-ACCACCTGGCTCATGGCGA-3') produjo un amplicón esperado de 165 nt, cuya secuencia fue confirmada por secuenciación de Sanger (100% de identidad de nt con la secuencia HTS).
Análisis posteriores sobre 64 árboles mostraron que 17 de ellos estaban infectados por PeSV. PeSV no se detectó en plantas asintomáticas. La mayor parte de árboles sintomáticos dieron positivo para PeSV aunque en alguno de ellos no se detectó por el método desarrollado lo que pone en evidencia la necesidad de mejorar los métodos de detección de este virus emergente en un cultivo de importancia en España como es el granado.

Para la primera descripción del granado como hospedado de PeSV y visualización con más detalle de la sintomatología producida por este virus se puede consultar la comunicación del 25 de noviembre de 2020.
Primeras detecciones de plum bark necrosis stem pitting associated virus en cerezo dulce en España (Autora: A.B. Ruiz-García)
11/06/21 18:11 ..... Divulgacion
En prospecciones en un area de cultivo de cerezo se analizaron muestras para estudiar el estado sanitario del cerezo. En el marco de esta prospección, se analizó una muestra (P7) de un cerezo dulce (Prunus avium), del cultivar Planera, que mostraba enrojecimiento y manchas necróticas en las hojas y se analizó mediante secuenciación masiva (HTS) con la plataforma NextSeq 500. El análisis de datos se realizó utilizando el software CLC Genomics Workbench 10.1.1 y Geneious 9.1.8. Después del control de calidad realizado por el software CLC, la muestra P7 rindió 42.683.262 lecturas. El ensamblado de novo produjo un total de 6.270 contigs (tamaño promedio de 1.884 nt). Los contigs posteriormente se analizaron por BLASTN y BLASTX. Este análisis mostró que ocho contigs estaban relacionados con el virus plum bark necrosis stem pitting associated virus (PBNSPaV). Tabién se detectó la presencia del virus cherry virus A representado con la presencia de 11 contigs.
Se realizó un análisis adicional mediante el mapeo de las lecturas contra todas las secuencias completas de PBNSPaV disponibles en las bases de datos utilizando el software Geneious. Los mejores resultados de mapeo se obtuvieron usando el aislado Pair-2 (KC590345) de Francia como referencia, lo que permitió la recuperación de una secuencia de 14.199 nt, que representa el genoma casi completo del aislado P7 (número de acceso de GenBank MN228561).
La identidad de nucleótidos entre el aislado P7 y el Par-2 fue del 99,27 %. Para confirmar la presencia de PBNSPaV en la muestra P7, se realizó la amplificación de una región parcial del gen CP por RT-PCR utilizando los cebadores específicos PBN-CP-F y PBN-CP-R. El producto de PCR de 301 pb obtenido (MN240523) fue secuenciado por Sanger y confirmó con 100% de identidad de la secuencia P7 recuperada por HTS (excluyendo los cebadores usados para la amplificación).
Un total de 24 muestras recogidas de la misma zona de cultivo de cerezo se analizaron mediante RT-PCR utilizando los mismos cebadores. Siete de estas muestras dieron positivo para PBNSPaV, lo que confirma aún más la presencia del virus en cerezp dulce en España. Este es el primer informe de PBNSPaV infectando cerezo dulce en España, lo que contribuye a una mejor comprensión de la epidemiología y la distribución del rango de huéspedes de este patógeno.
Se realizó un análisis adicional mediante el mapeo de las lecturas contra todas las secuencias completas de PBNSPaV disponibles en las bases de datos utilizando el software Geneious. Los mejores resultados de mapeo se obtuvieron usando el aislado Pair-2 (KC590345) de Francia como referencia, lo que permitió la recuperación de una secuencia de 14.199 nt, que representa el genoma casi completo del aislado P7 (número de acceso de GenBank MN228561).
La identidad de nucleótidos entre el aislado P7 y el Par-2 fue del 99,27 %. Para confirmar la presencia de PBNSPaV en la muestra P7, se realizó la amplificación de una región parcial del gen CP por RT-PCR utilizando los cebadores específicos PBN-CP-F y PBN-CP-R. El producto de PCR de 301 pb obtenido (MN240523) fue secuenciado por Sanger y confirmó con 100% de identidad de la secuencia P7 recuperada por HTS (excluyendo los cebadores usados para la amplificación).
Un total de 24 muestras recogidas de la misma zona de cultivo de cerezo se analizaron mediante RT-PCR utilizando los mismos cebadores. Siete de estas muestras dieron positivo para PBNSPaV, lo que confirma aún más la presencia del virus en cerezp dulce en España. Este es el primer informe de PBNSPaV infectando cerezo dulce en España, lo que contribuye a una mejor comprensión de la epidemiología y la distribución del rango de huéspedes de este patógeno.
Actualización sobre las especies de virus que infectan vid (Autora: A.B. Ruiz-García)
26/04/21 20:05 ..... Divulgacion
Hasta la fecha, se han detectado 86 virus que infectan vides de todo el mundo. Algunos de estos virus se asocian con enfermedades económicamente perjudiciales y están asociados con los cuatro principales complejos de enfermedades conocidos como degeneración infecciosa, enrollado, madera rugosa y jaspeado.
A continuación una actualización de los virus que infectan vid:
virusvidAvirusvidB
A continuación una actualización de los virus que infectan vid:
virusvidAvirusvidB
Recordando que el virus de los cítricos Citrus tatter feaf ha sido detectado por primera vez en Europa (Autora: A.B. Ruiz-García)
12/03/21 21:55 ..... Divulgacion
El virus de la hoja de cítricos Citrus tatter leaf virus (CTLV) es un capillovirus que pertenece a la familia Betaflexiviridae que se transmite de forma mecánica aunque no se han identificado todavía vectores.
Actualmente la ICTV lo considera como una cepa de un virus conocido de frutales, el Apple stem grooving virus (ASGV).
La mayoría de las variedades de cítricos comerciales infectadas son asintomáticas excepto cuando se propagan sobre patrones Poncirus trifoliata o Poncirus trifoliata x Citrus sinensis. Es entonces cuando los árboles presentan enanismo, hojas cloróticas, incompatibilidad en la unión del injerto lo que conlleva a al deterioro final del árbol. Esto supone un problema grave porque los patrones basados en Poncirus trifoliata y sus híbridos se utilizan ampliamente en todas las zonas productoras del mundo por su tolerancia al virus de la tristeza de los cítricos y a las especies de Phytophthora.
Este virus se describió por primera vez en California y posteriormente en Australia, Korea, Nigeria, Japan, Sudafrica, y China.
Sin embargo en el año 2019 (Turgut Alas et al., Saudi Journal of Biological Sciences 26, 995-998) detectaron este virus Chipre, siendo la primera detección del este virus en cítricos en Europa.
La sintomatología que los autores observaron fue la siguiente:

Distorsión de hojas, necrosis y síntomas cloróticos en hojas infectadas con CTLV (T. Alas et al. 2019)
Para la identificación del virus emplearon RT-PCR convencional empleando los iniciadores específicos:
TL1F: TGAAAACCTTTGCTGCCACCTCT
TL1R: TACTCTCCGAACCTGCCTCGAAA
que amplificaba un fragmento de 309 pb a una temperatura de annealing de 59ºC
Se trata pues de una detección muy a tener en cuenta puesto que este virus se ha considerado hasta la fecha como de cuarentena para la citricultura española.
Actualmente la ICTV lo considera como una cepa de un virus conocido de frutales, el Apple stem grooving virus (ASGV).
La mayoría de las variedades de cítricos comerciales infectadas son asintomáticas excepto cuando se propagan sobre patrones Poncirus trifoliata o Poncirus trifoliata x Citrus sinensis. Es entonces cuando los árboles presentan enanismo, hojas cloróticas, incompatibilidad en la unión del injerto lo que conlleva a al deterioro final del árbol. Esto supone un problema grave porque los patrones basados en Poncirus trifoliata y sus híbridos se utilizan ampliamente en todas las zonas productoras del mundo por su tolerancia al virus de la tristeza de los cítricos y a las especies de Phytophthora.
Este virus se describió por primera vez en California y posteriormente en Australia, Korea, Nigeria, Japan, Sudafrica, y China.
Sin embargo en el año 2019 (Turgut Alas et al., Saudi Journal of Biological Sciences 26, 995-998) detectaron este virus Chipre, siendo la primera detección del este virus en cítricos en Europa.
La sintomatología que los autores observaron fue la siguiente:

Distorsión de hojas, necrosis y síntomas cloróticos en hojas infectadas con CTLV (T. Alas et al. 2019)
Para la identificación del virus emplearon RT-PCR convencional empleando los iniciadores específicos:
TL1F: TGAAAACCTTTGCTGCCACCTCT
TL1R: TACTCTCCGAACCTGCCTCGAAA
que amplificaba un fragmento de 309 pb a una temperatura de annealing de 59ºC
Se trata pues de una detección muy a tener en cuenta puesto que este virus se ha considerado hasta la fecha como de cuarentena para la citricultura española.
¿Qué son los aislados Resistance-Breaking (RB) del virus de la tristeza de los cítricos? (Autora: A.B. Ruiz-García)
05/02/21 12:33 ..... Divulgacion
Tener controlada mediante patrones tolerantes la enfermedad de la tristeza de los cítricos causada por el virus CTV y que ha producido la muerte de más de 50 millones de árboles sólo en España, no quiere decir que debamos olvidar esta problemática.
Los virus evolucionan, se adaptan, mutan y recombinan para vencer resistencias de hospedadores y avanzar en su objetivo, su supervivencia. Y así han aparecido y se han descrito nuevos grupos o genotipos de CTV, como el caso de los aislados Resistance Breaking (RB) formados por un grupo de aislados con características genéticas y biológicas particulares, que se han dividido en dos genotipos, los del grupo 1 RB y los del grupo 2 RB.
Los aislados RB, a diferencia del resto de aislados del virus de la tristeza, pueden replicarse y moverse de forma sistémica en Poncirus trifoliata, que muestra resistencia a otros aislados de CTV.
Estos genotipos RB, se han descrito en Nueva Zelanda, República Dominicana, Puerto Rico, Sudáfrica, California, Brasil y Marruecos.
En España solo existen aislados poco agresivos de CTV, que producen tristeza cuando naranjos dulces, mandarinos, limas y pomelos se injertan sobre naranjo amargo, y se produce una incompatibilidad entre patrón y variedad, por lo que se mantiene como prioridad evitar la entrada de CTV no europeos que incluyen ahora los aislados RB para preservar nuestra citricultura.
Se han diseñado y validado los iniciadores específicos siguientes que permiten realizar PCRs convencionales. El protocolo de detección es el siguiente:
Muestreo: 5 brotes terminales (10 cm) alrededor de la copa del árbol
Purificación de ácidos nucleicos: RNeasy Plant kit o similar
Preparación del cóctel:
Para cada reacción de 25 microlitros (3 microlitros de RNA purificado)
H2O 10,7 microlitros
Tampón 5x Promega 5 microlitros
Cl2Mg 25 mM (uso 1,5 mM) 1,5 microlitros
dNTP (2,5 mM cada uno) uso 0,25 mM 2,5 microlitros
Iniciador F1 (25 microM) uso 1microM 1 microlitro
Inciador R1 (25 microM) uso 1microM 1 microlitro
Promega AMV (10U/microlitro) 0,1 microlitros
GoTaq G2 Promega (5 U/microlitro) 0,2 microlitros
Según el genotipo se seleccionan los iniciadores:
RB group1
Iniciador F1 AGT GGT GGA GAT TAC GTT G
Inciador R1 TAC ACG CGA CAA ATC GAG
RB group 2
Iniciador F1 CGG AAG GGA CTA CGT GGT
Iniciador R1 CGT TTG CAC GGG TTC AAT G
Condiciones del Termociclador
40 min -----------------42ºC
4 min ------------------- 94ºC
40 ciclos:
30 s ---------------------- 92ºC
30 s ---------------------- 60ºC
1 min -------------------- 72ºC
10 min ----------------- 72ºC
 orcid.org/0000-0001-8406-7963
orcid.org/0000-0001-8406-7963