Actualización sobre las especies de virus que infectan vid
A continuación una actualización de los virus que infectan vid:
virusvidAvirusvidB
Recordando que el virus de los cítricos Citrus tatter feaf ha sido detectado por primera vez en Europa
Actualmente la ICTV lo considera como una cepa de un virus conocido de frutales, el Apple stem grooving virus (ASGV).
La mayoría de las variedades de cítricos comerciales infectadas son asintomáticas excepto cuando se propagan sobre patrones Poncirus trifoliata o Poncirus trifoliata x Citrus sinensis. Es entonces cuando los árboles presentan enanismo, hojas cloróticas, incompatibilidad en la unión del injerto lo que conlleva a al deterioro final del árbol. Esto supone un problema grave porque los patrones basados en Poncirus trifoliata y sus híbridos se utilizan ampliamente en todas las zonas productoras del mundo por su tolerancia al virus de la tristeza de los cítricos y a las especies de Phytophthora.
Este virus se describió por primera vez en California y posteriormente en Australia, Korea, Nigeria, Japan, Sudafrica, y China.
Sin embargo en el año 2019 (Turgut Alas et al., Saudi Journal of Biological Sciences 26, 995-998) detectaron este virus Chipre, siendo la primera detección del este virus en cítricos en Europa.
La sintomatología que los autores observaron fue la siguiente:

Distorsión de hojas, necrosis y síntomas cloróticos en hojas infectadas con CTLV (T. Alas et al. 2019)
Para la identificación del virus emplearon RT-PCR convencional empleando los iniciadores específicos:
TL1F: TGAAAACCTTTGCTGCCACCTCT
TL1R: TACTCTCCGAACCTGCCTCGAAA
que amplificaba un fragmento de 309 pb a una temperatura de annealing de 59ºC
Se trata pues de una detección muy a tener en cuenta puesto que este virus se ha considerado hasta la fecha como de cuarentena para la citricultura española.
¿Qué son los aislados Resistance-Breaking (RB) del virus de la tristeza de los cítricos?
Tener controlada mediante patrones tolerantes la enfermedad de la tristeza de los cítricos causada por el virus CTV y que ha producido la muerte de más de 50 millones de árboles sólo en España, no quiere decir que debamos olvidar esta problemática.
Los virus evolucionan, se adaptan, mutan y recombinan para vencer resistencias de hospedadores y avanzar en su objetivo, su supervivencia. Y así han aparecido y se han descrito nuevos grupos o genotipos de CTV, como el caso de los aislados Resistance Breaking (RB) formados por un grupo de aislados con características genéticas y biológicas particulares, que se han dividido en dos genotipos, los del grupo 1 RB y los del grupo 2 RB.
Los aislados RB, a diferencia del resto de aislados del virus de la tristeza, pueden replicarse y moverse de forma sistémica en Poncirus trifoliata, que muestra resistencia a otros aislados de CTV.
Estos genotipos RB, se han descrito en Nueva Zelanda, República Dominicana, Puerto Rico, Sudáfrica, California, Brasil y Marruecos.
En España solo existen aislados poco agresivos de CTV, que producen tristeza cuando naranjos dulces, mandarinos, limas y pomelos se injertan sobre naranjo amargo, y se produce una incompatibilidad entre patrón y variedad, por lo que se mantiene como prioridad evitar la entrada de CTV no europeos que incluyen ahora los aislados RB para preservar nuestra citricultura.
Se han diseñado y validado los iniciadores específicos siguientes que permiten realizar PCRs convencionales. El protocolo de detección es el siguiente:
Muestreo: 5 brotes terminales (10 cm) alrededor de la copa del árbol
Purificación de ácidos nucleicos: RNeasy Plant kit o similar
Preparación del cóctel:
Para cada reacción de 25 microlitros (3 microlitros de RNA purificado)
H2O 10,7 microlitros
Tampón 5x Promega 5 microlitros
Cl2Mg 25 mM (uso 1,5 mM) 1,5 microlitros
dNTP (2,5 mM cada uno) uso 0,25 mM 2,5 microlitros
Iniciador F1 (25 microM) uso 1microM 1 microlitro
Inciador R1 (25 microM) uso 1microM 1 microlitro
Promega AMV (10U/microlitro) 0,1 microlitros
GoTaq G2 Promega (5 U/microlitro) 0,2 microlitros
Según el genotipo se seleccionan los iniciadores:
RB group1
Iniciador F1 AGT GGT GGA GAT TAC GTT G
Inciador R1 TAC ACG CGA CAA ATC GAG
RB group 2
Iniciador F1 CGG AAG GGA CTA CGT GGT
Iniciador R1 CGT TTG CAC GGG TTC AAT G
Condiciones del Termociclador
40 min -----------------42ºC
4 min ------------------- 94ºC
40 ciclos:
30 s ---------------------- 92ºC
30 s ---------------------- 60ºC
1 min -------------------- 72ºC
10 min ----------------- 72ºC
Apple stem pitting virus, un virus de amplia distribución y que infecta diversas especies vegetales
En un estudio realizado en nuestro laboratorio en una prospección de níspero en la región de Segorbe, hemos detectado unas variantes capaces de infectar el níspero aunque sin producir sintomatología.
Para avanzar en el conocimiento y poder disponer de herramientas de diagnóstico de estas variantes de níspero, se ha secuenciado completamente un aislado y caracterizamos parcialmente 10 aislados más para ver filogenéticamente donde se situaban.
La estructura del aislado de níspero muestra que mantiene los cinco ORFs (RdRP, TGB1, TGB2, TGB3 y CP) como se muestra en la figura
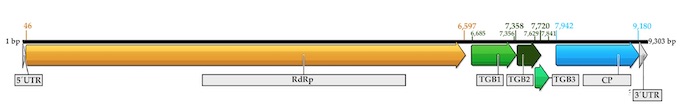
El análisis filogenético del aislado completo de ASPV lo sitúa próximo a las variantes de peral en color verde.
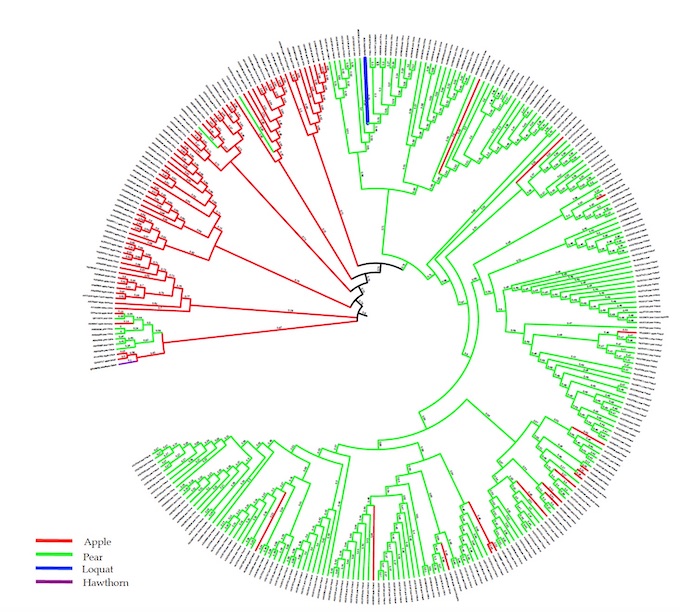
El níspero se confirma como nuevo huésped de ASPV y se han diseñado iniciadores adaptados a la secuencia del aislado del níspero que ha permitido evaluar la prevalencia en Segorbe que ha resultado ser del 15%.
Un misterio resuelto, el virus asociado al amarilleamiento de la hoja del olivo
Estudios posteriores sólo avanzaron parcialmente en su conocimiento del genoma, ya que únicamente se logró conocer alrededor de 5000 nucleótidos. Con este conocimiento se diseñaron técnicas de diagnóstico y se averiguó que OLYaV era uno de los virus del olivo más extendidos en muchas zonas donde se cultivaba. Así, se descubrieron diferentes niveles de incidencia: California (EE. UU.) (93%), Italia (64-21%), Túnez (49%), Líbano (24%), Siria (15%), Grecia (5%) y Albania (2%).
Sin embargo OLYaV permanecía como una especie no asignada en la familia Closteroviridae porque se necesitaban más datos biológicos y moleculares para su clasificación.
En un estudio que recientemente hemos publicado, hemos descubierto la secuencia completa de OYLaV de 16700 nucleótidos, incluyendo todo su potencial de codificación.
En la figura se muestra la organización genómica de OLYaV. L-Pro: proteasa leader papain-like; Met-T: dominio metal transferasa; Helicase: dominio de la helicasa viral; RdRp: polimerasa RNA dependiente; Las flechas azules indican la posición de corte del proteasa.
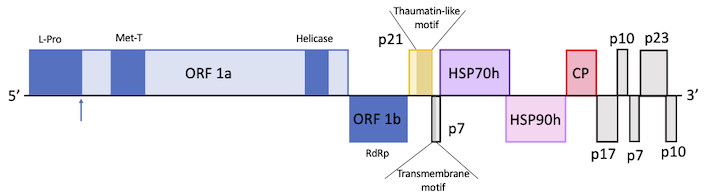
La secuencia completa muestra un genoma con estructura típica de la familia Closteroviridae. Contiene 11 pautas de lectura abiertas (ORF,) entre ellos el complejo ORF1ab que codifica para los tres dominios conservados típicos, proteasa líder similar a la papaína, metiltransferasa y helicasa y además la RdRp, además de los genes de las proteínas HSP70 y HSP90.
OLYaV exhibe dos características genómicas originales. Primero, la ORF2 codifica una proteína similar a la taumatina y segundo, no codifica una proteína CPm. Curiosamente, estas dos características genómicas son compartidas por otras dos especies de Closteroviridae no asignadas, PVB and AV1. Además el análisis filogenético, fundamentalmente el estudio de los genes comunes en todas las especies de closterovirus (ORF1a, ORF1b (RdRp), HSP70, HSP90 y CP) ha abierto nuevas posibilidades en la clasificación taxonómica de OLYaV y del persimmon virus B (PVB) y el actidinia virus 1 (AV1), lo que sugiere de forma consistente que forman un nuevo género dentro de la familia Closteroviridae.
Primera detección del Grapevine asteroid mosaic associated virus en viñas de Tempranillo y Macabeo en España
Además de estos se descubrió la presencia del virus asteroide de la vid y la secuencia completa se recuperó siendo de 6692 nucletótidos. El análisis filogenético mostró que el aislado español se situaba en un clado diferente a los que previamente estaban descritos, como se muestra en la figura.
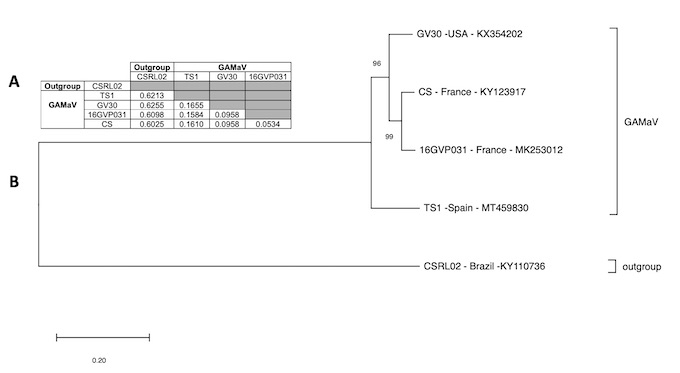
Basándonos en la secuencia del aislado TS1 diseñamos dos iniciadores - GAMaV-6010F, 5’CCCTCCTCCTAGCGACGACC3’ y GAMaV- 6426R, 5’GGGTTGAGACGGCGGAGATC3’ - que amplifican un fragmento de 417 nt en la región de la cáspida y que ha permitido detectar los aislados españoles con fiabilidad, revelando una incidencia en el area estudiada de casi el 10%, puesto que los análisis con los iniciadores descritos por otros autores no detectaban los españoles. Estas han sido las primeras detecciones en España de este virus.
Clasificación de virus vegetales de doble cadena de RNA
Familia Amalgaviridae
- Género
- Amalgavirus
Familia Partitiviridae
- Géneros
- Alphapartitivirus
- Betapartitivirus
- Deltapartitivirus
Familia Reoviridae
Subfamilia Spinareovirinae
- Géneros
- Fijivirus
- Oryzavirus
Subfamilia Sedoreovirinae
- Género
- Phytoreovirus
Muestreo para el virus de la sharka
Se han de tomar si es posible muestras con síntomas típicos: flores, hojas y frutos que presenten los síntomas
En caso de ser muestras sin síntomas: brotes de por lo menos un año con hojas maduras o completamente desarrolladas de la parte central de cada una de las ramas principales (la detección no es fiable en brotes de menos de un año). Las muestras deberían tomarse, como mínimo, en cuatro sitios diferentes (p. ej., cuatro ramas o cuatro hojas) de cada planta; esto es indispensable ya que la distribución del PPV es desigual
El muestreo no debería realizarse durante los meses de temperaturas más altas
Las pruebas son menos fiables si se realizan con muestras tomadas durante el otoño que con las obtenidas al principio de la primavera
En primavera, las muestras pueden ser flores, brotes con las hojas totalmente desarrolladas o frutos.
En verano y en otoño pueden utilizarse para el análisis las hojas maduras y la piel de los frutos maduros recogidos del campo o de los lugares de embalaje.
Las flores, las hojas, los brotes y la piel del fruto pueden almacenarse a una temperatura de 4ºC por no más de 10 días antes del procesamiento.
Los frutos pueden almacenarse durante un mes a una temperatura de 4ºC antes del procesamiento.
En invierno pueden seleccionarse las yemas dormidas o los tejidos de corteza de la zona basal de ramillas, brotes y ramas, o espolones enteros.
La psorosis de los cítricos severa transmisible B
La psoriasis severa transmisible o Psoriasis B se encuentra en Argentina y en Uruguay y está ausente en la región EPPO.
La transmisión se ha asociado por un lado al hongo Olpidoium brassiacae
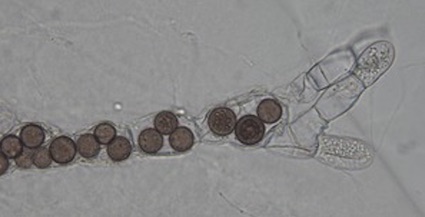
Síntomas
Presencia de descamaciones en la corteza del tronco y ramas
Los síntomas se inician cuando el árbol tiene de 10-15 años con pequeñas lesiones en la corteza
Las lesiones se levantan, se secan y se van desprendiendo
La psorosis B causa descamamiento exuberante en ramas delgadas de los árboles incluso en ramas de menos de 1cm de diámetro

Otras características son que algunas lesiones exudan goma en los límites de las zonas descamadas y en corte transversal de ramas afectadas en la madera se observan zonas de color pardo y de forma irregular que son exudaciones de goma que invaden los vasos de xilema.

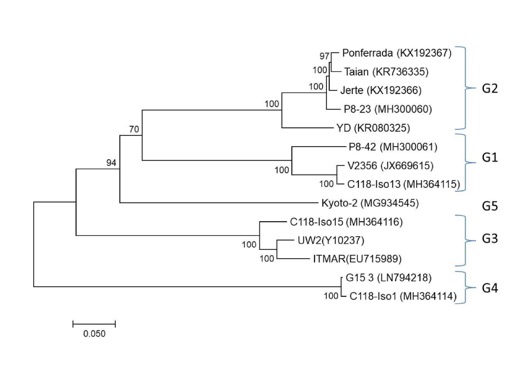
Primeras infecciones mixtas en cerezo por diferentes genotipos del virus de la cereza pequeña


Este tipo de infecciones mixtas en cerezo con diferentes genotipos de LChV-1 no habían sido descritas hasta la fecha. Ya que no existe vector conocido del virus, la coexistencia de estas variantes podría ser atribuida a prácticas agronómicas como el injerto. Estas infecciones mixtas pueden tener implicaciones en la patogenicidad del virus ya que pueden además producirse recombinaciones. La presencia de dos genotipos diferentes además sugiere dos eventos diferentes de introducción en España.
En un estudio que hemos realizado en colaboración con investigadores de Francia, Grecia y Eslovaquia, hemos logrado un avance en el conocimiento de la diversidad del virus ampliando las cuatro agrupaciones o “clusters” que se conocían a cinco. Estos avances permiten el diseño de nuevas herramientas de diagnóstico capaces de detectar todas las variantes de LChV-1 conocidas hasta el momento.
En la figura de abajo se muestra un árbol filogenético (Maximum likelihood; 500 réplicas) que relaciona las secuencias completas disponibles hasta la fecha del LChV-1.
De vuelta con el Grapevine pinot gris virus, y los que no disponen de PCR a tiempo real?
Pues con una RT-PCR convencional y para ello os indicamos unos iniciadores que amplifican diferentes regiones del genoma del virus.
Nuestra recomendación, al menos utilizar dos PCRs diferentes, porque este virus es variable y así disminuimos posibilidades de falsos negativos.
Iniciadores de Beuve et al.:
Proteína de movimiento (770pb)
Pg-Mer-F1 5′-GGAGTTGCCTTCGTTTACGA-3′
Pg-Mer-R1 5′-GTACTTGATTCGCCTC GCTCA-3′
Iniciadores de Glasa et al.:
5’UTR (618pb)
GPG-14F 5’-AATTGATCCCGTGTAGTGC-3’
GPG-632R 5’-TCCGAGGACGATGAACCTC-3’
Proteína de movimiento (302pb)
GPG-5637F 5’-ATTGCGGAGTTGCCTTCAAG-3’
GPG-5939R 5’-CTGAGAAGCATTGTCCCATC-3’
Proteína de cápside (411pb)
GPG-6609F 5’-GAGATCAACAGTCAGGAGAG-3’
GPG-7020R 5’-GACTTCTGGTGCCTTATCAC-3’
El protocolo para su detección por RT-PCR convencional es el siguiente:
Muestreo: 4 brotes terminales (10 cm) alrededor de la cepa
Purificación de ácidos nucleicos: RNeasy Plant kit o similar
Preparación del cóctel: Para cada reacción de 25 microlitros (3 microlitros de RNA purificado)
H2O 10,7 microlitros
Tampón 5x Promega 5 microlitros
Cl2Mg 25 mM (uso 1,5 mM) 1,5 microlitros
dNTP (2,5 mM cada uno) uso 0,25 mM 2,5 microlitros
Iniciador 1 (25 microM) uso 1microM 1 microlitro
Iniciador 2 (25 microM) uso 1microM 1 microlitros
GoTaq G2 Promega (5 U/microlitro) 0,2 microlitros
AMV Promega (10 U/microlitro) 0,1 microlitro
Condiciones del Termociclador
30 min ------------------ 45ºC
4 min ------------------- 94ºC
40 ciclos: 30 s ---------------------- 92ºC
30 s ---------------------- 50ºC
1 min -------------------- 72ºC
10 min ----------------- 72ºC
Un virus emergente en vid, Grapevine pinot gris virus
Desde su primera descripción en Italia, el virus se ha descrito en otros países como Grecia, República Checa, Eslovaquia, Eslovenia, Francia, Alemania, Portugal, Corea del Sur, China, Canadá, EEUU, Turquía, Brasil, Croacia, Rumanía, Ucrania o Australia.
En España nuestro grupo de investigación describió por primera su presencia en el país en el año 2017, infectando variedades de Garnacha, Bobal y Tempranillo. Con el objetivo de comprender y mejorar la epidemiología de la enfermedad y desarrollar estrategias de control hemos desarrollado una RT-PCR a tiempo real que permite la detección y cuantificación del virus, con un limite de 70 copias del virus en material vegetal. También se ha aplicado a la detección en un posible vector Colomerus vitis.

Además hemos descubierto un nuevo polimorfismo en el genoma del virus que hace que la proteína de movimiento sea más pequeña.
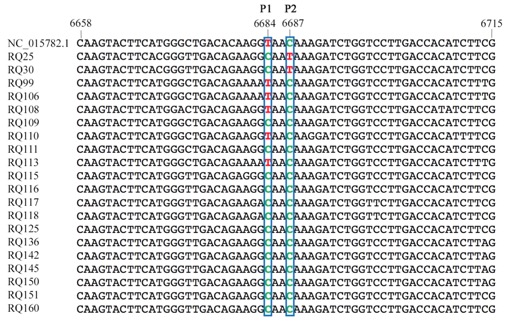
Un estudio filogenético basado en esta región genómica muestra una alta variaibilidad entre los aislados españoles, que se localizan en diversos grupos o “clusters” independientemente de tener o no polimorfismos en la proteína de movimiento (nuevo polimorfismo, círculo rojo completo; polimorfismo previamente descrito por un grupo italiano, círculo con sólo la circunferencia roja)
El desarrollo de esta metodología de detección y el avance en la diversidad del virus, permitirá mejorar herramientas de control y diagnóstico del virus.
La importancia de los insectos vectores en la dipersión de los virus
¿Qué hace interesantes los virus vegetales?
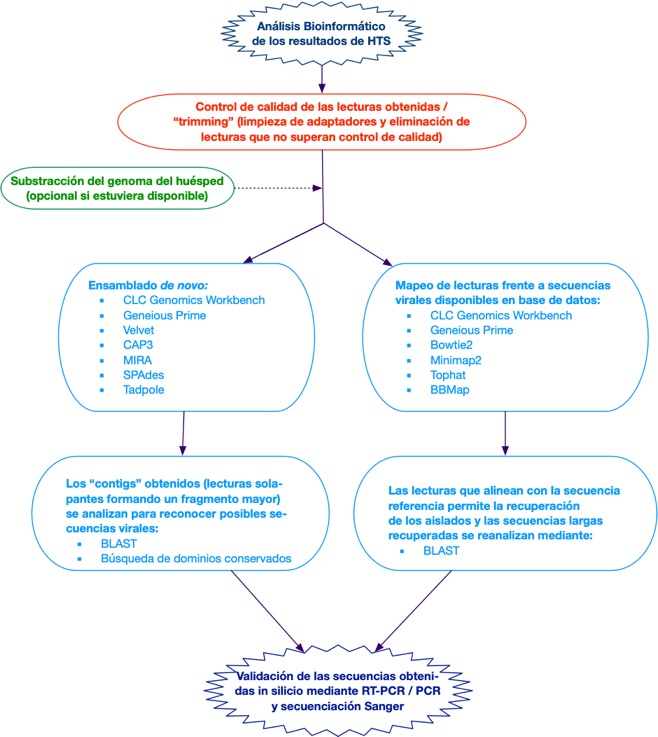
¿Cuál es el esquema del análisis bioinformático de los resultados de secuenciación masiva para el estudio de virus?
Los viroides ¿qué son?
Su genoma es una molécula de RNA monocatenario circular de 270 a 470 nucleótidos, que no está encapsidada y no es codificante. No tienen pues proteínas propias y son parásitos obligados de las células de la planta huésped.
Se replican de forma autónoma utilizando el sistema de transcripción de las células infectadas. Siguen el modelo de realización del círculo rodante y está relacionado con la naturaleza circular de los viroides.
Hay dos familias de viroides:
Pospiviroidae que se replican en el núcleo
Avsunviroidae que se replican en los cloroplastos
Los viroides invaden la planta huésped por una herida inicial y movimiento célula a célula via plasmodesmos e invasión a larga distancia por los tejidos conductores del floema
La transmisión planta a planta es:
- principalmente mecánica
- pocos casos por pulgones, cuando una molécula de viroide se encapsida en partículas de virus en confección virus-viroide
- por multiplicación vegetativa
- semilla
Los síntomas son similares a los virus fitopatógenos, raquitismo, epinastia, decoloraciones foliares, aclarado de nervios, necrosis, distorsión de hojas, lesiones necróticas locales o generalizadas
El primer viroide descubierto fue el causante de la enfermedad del tubérculo fusiforme de la patata Potato splindle tuber viroid o PSTVd
El segundo viroide fue el de la exocortis de los cítricos, citrus exocortis viroid o CEVd

otro viroide de importancia es el Peach latente mosaic viroid o PLMVd que tiene una sintomatología muy característica en los frutos
Clasificación de virus vegetales de DNA de cadena simple
Los más representativos se clasifican en los siguientes géneros
Familia Geminiviridae
Géneros:
* Becurtovirus
* Begomovirus
* Capulavirus
* Curtovirus
* Grablovirus
* Eragrovirus
* Mastrevirus
* Topocuvirus
* Turncurtovirus
Familia Nanoviridae
Géneros:
* Babuvirus
* Nanovirus
¿Cómo evitar la introducción de cepas agresivas tipo M de sharka?
temprana de melocotonero en amplias zonas de un país como España que
produce más de 700.000 toneladas de melocotones.
Hay unas medidas importantes que deben conocerse y aplicarse para evitar o reducir los riesgos de
introducción de tipos agresivos del virus de la sharka:
- La más básica: No introducir material vegetal de zonas donde exista PPV M, evitando la toma de varetas de material vegetal especialmente si son recolectadas en el sur de Francia, Italia, Grecia o cualquier país de Europa del Este.
- Una medida lógica: No plantar material vegetal de frutales de hueso sin un análisis previo de PPV y que las plantaciones se realicen con material certificado libre de PPV
- Una medida de atención: Vigilar los árboles en floración, durante el engrosamiento del fruto y en el momento de la recolección y observar si aparecieran síntomas
- Una medida más: Analizar los melocotoneros que presenten síntomas de PPV
¿Por qué no es suficiente con la sensibilidad y la especificidad en el diagnóstico?
Fijémonos en la tabla de contingencia de variables de diagnóstico:
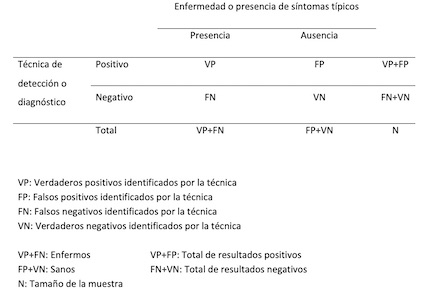
La sensibilidad de la prueba diagnóstica se define como la probabilidad de que el resultado de la prueba sea positivo en una planta enferma. Representa la proporción de verdaderos positivos diagnosticados por la técnica entre las plantas enfermas VP/(VP+FN)
La especificidad de la prueba diagnóstica se define como la probabilidad de que el resultado de la prueba sea negativo en una planta sana. Representa el porcentaje de resultados negativos respecto del total de plantas sanas VN/(FP+VN)
La sensibilidad no contempla los falsos positivos y la especificidad no contempla los falsos negativos que rinde la técnica. Y es precisamente esto lo que necesita conocer el técnico o lo que es lo mismo saber ¿cúal es la probabilidad de que un resultado positivo por la técnica sea un verdadero positivo y no un falso positivo, independientemente de su sensibilidad? y ¿cúal es la probabilidad de que un resultado negativo por la técnica sea un verdadero negativo y no un falso negativo, independientemente de su especificidad? La respuesta la dan los valores predictivos positivo y negativo, que se calculan considerando las filas de la tabla de contingencia al contrario que la sensibilidad y especificidad que se calculan en las columnas de la tabla de contingencia.
El valor predictivo positivo de una prueba diagnóstica se define como la probabilidad de que la planta esté enferma habiendo dado positivo en la prueba. Así pues, el valor predictivo positivo representa el porcentaje de plantas realmente enfermas respecto del total de plantas que han dado positivo VP/(VP+FP). Un valor alto indica que la probabilidad de que la planta esté realmente enferma habiendo dado positivo en la prueba diagnóstica es muy alta.
El valor predictivo negativo se define como la probabilidad de que una planta esté sana habiendo dado negativo. Representa el porcentaje de plantas sanas respecto del total de plantas que han dado negativo en la prueba VN/(VN+FN).
Sin embargo, si aplicamos el teorema de Bayes se puede concluir que los valores predictivos dependen de la prevalencia de la enfermedad que tratamos de diagnosticar según la siguiente relación: valor predictivo positivo= (sensibilidad de la prueba x prevalencia de la enfermedad) / ((sensibilidad x prevalencia) + [(1 - especificidad) x (1 - prevalencia)]). Como puede observarse, cuanto mayor sea la prevalencia de la enfermedad en la población mayor será el valor predictivo positivo de la prueba diagnóstica, aún manteniéndose constantes su sensibilidad y especificidad; de forma similar ocurre con el valor productivo negativo.
Así pues, si sensibilidad y especificidad no dan una respuesta apropiada por no contemplar falsos positivos y falsos negativos respectivamente y los valores predictivos positivo y negativo varían con la prevalencia ¿qué parámetros son los apropiados para evaluar la técnica diagnóstica? La respuesta la dan “likelihood ratios” de positivo y negativo o razón de verosimilitud. Así la razón de verosimilitud positiva es la proporción de verdaderos positivos correctamente identificados por la técnica, o sea, la sensibilidad dividido por la proporción de falsos positivos que la técnica diagnostica FP/VP+FP, o lo que es lo mismo 1-especificidad = 1-(VN/(VN+FP)).
La razón de verosimilitud negativa es la proporción de falsos negativos que la técnica diagnostica (1-sensibilidad), dividido por la proporción de verdaderos negativos correctamente identificados por la técnica o lo que es lo mismo, la especificidad
La enorme ventaja de emplear las razones de verosimilitud es que pueden usarse para cuantificar la probabilidad de la enfermedad para una planta individual. El teorema de Bayes se usa para traducir la información suministrada por los las razones de verosimilitud y probabilidad de enfermedad. El teorema de Bayes establece que la razón de probabilidades o razón de momios pre-test, que es lo mismo el cociente de la probabilidad de enfermedad y la probabilidad de no enfermedad antes de realizar el test, multiplicado por la razón de verosimilitud da lugar a la razón de probabilidades post-test de la enfermedad o sea el cociente de la probabilidad de la enfermedad y la probabilidad de no enfermedad después de realizar el análisis. La probabilidad post-test se puede calcular de la siguiente forma:
Probabilidad Pre-test = Prevalencia
Razón de probabilidades Pre-test = Prevalencia/(1-Prevalencia)
Razón de probabilidades Post-test = Razón de probabilidades Pre-test x Razón de verosimilitud
Probabilidad Post-test = Razón de probabilidades Post-test /(1+ Razón de probabilidades Post-test)
Además las razones de verosimilitud de diversos métodos pueden combinarse en la formula de modo que: Razón de probabilidades Post-test = Razón de probabilidades Pre-test x Razón de verosimilitud 1 x Razón de verosimilitud 2 x Razón de verosmilitud 3.
Así pues la probabilidad pre-test de enfermedad puede compararse con la probabilidad post-test. La diferencia entre la previa y la posterior es una manera muy efectiva de analizar la eficiencia de un método diagnóstico.
El enanismo de la satsuma

El virus se transmite rápido por injerto entre cítricos y mecánicamente entre cítricos y huéspedes no cítricos. Su dispersión natural parece ser que es por vectores del suelo en las comarcas de Japón y también se ha indicado una posible transmisión entre Viburnum odoratissimum y cítricos.
La enfermedad produce raquitismo y las hojas se abarquillan y adquieren forma de cuchara.
Los frutos son de baja calidad y bajo valor comercial y es una amenaza para los países productores.
El enanismo de la satsuma severa es un problema en Japón.
 orcid.org/0000-0001-8406-7963
orcid.org/0000-0001-8406-7963